|
|||
|
|||
以下の情報は,佐藤行人さんから教えていただきました.
をチェックします.その後,Results を押して次に進みます. |
|||
|
|||
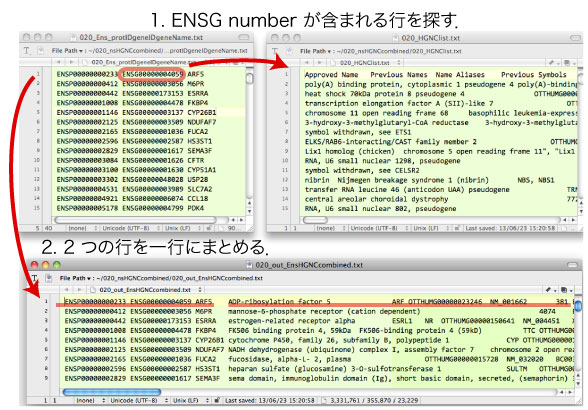
Ensembl の BioMart から得られたファイル (左: 020_Ens_protIDgeneIDgeneName.txt) と HGNC から得られたファイル (右: 020_HGNClist.txt) 間で,同じ ENSG ID が含まれる行を合わせるスクリプトです (2013 年 6 月).
|
|||
 |
|||
|
|||
|
|||
以下の情報は,佐藤行人さんから教えていただきました.
をチェックします.その後,Results を押して次に進みます. |
|||
|
|||
Ensembl の BioMart から得られたファイル (左: 020_Ens_protIDgeneIDgeneName.txt) と HGNC から得られたファイル (右: 020_HGNClist.txt) 間で,同じ ENSG ID が含まれる行を合わせるスクリプトです (2013 年 6 月).
|
|||
 |
|||