|
|
| 2016 年 3 月 19 日 改訂 |
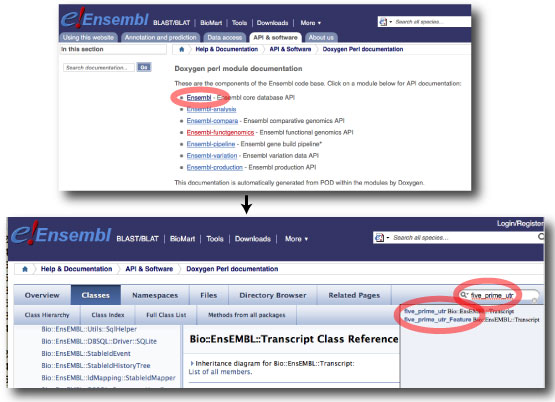
Ensembl API は,Ensembl のデータ取得行う方法の一つです.Perl のモジュール群 (スクリプトの集まり) を使います.インストールの手順はこちらに書きました.
|
|
以下のコマンドを,実行してください. versionCheck.pl.tar.gz からダウンロード&解凍できます.
use Bio::EnsEMBL::ApiVersion;
printf( "The API version used is %s\n", software_version() );
|
|
まずは,「Ensembl API を極める:1-2. データベースに接続する」とこれ以降にある例題を一通りやった方が良いです.
|
|
#!/usr/bin/perl
use strict;
use warnings;
use Bio::EnsEMBL::Registry;
use Bio::EnsEMBL::ApiVersion;
print "Ens", software_version(), "\n";
my $registry = 'Bio::EnsEMBL::Registry';
$registry->load_registry_from_db(
-host => asiadb.ensembl.org',
-user => 'anonymous'
);
my @db_adaptors = @{ $registry->get_all_DBAdaptors() };
foreach my $db_adaptor (@db_adaptors) {
my $db_connection = $db_adaptor->dbc();
printf(
"species/group\t%s/%s\ndatabase\t%s\nhost:port\t%s:%s\n\n",
$db_adaptor->species(), $db_adaptor->group(),
$db_connection->dbname(), $db_connection->host(),
$db_connection->port()
);
}
|
ミラーサイトに接続
Ensembl API を用いる場合は,常に黒字の部分が必要です.
-host => 'asiadb.ensembl.org',
の部分はアジアのミラーサイトに接続を意味します.ヨーロッパのサイトは,
-host => 'ensembldb.ensembl.org',
です. |
途中でスクリプトが止まる
ヨーロッパのサイトからたくさんの遺伝子を処理する場合など,ネット接続の問題で途中でスクリプトがストップする場合があります.
[junINOUEb:Downloads]$ perl homology_compara.pl
The API version used is 84
Query geneID: ENSG00000157601
UNIVERSAL->import is deprecated and will be removed in a future perl at
/Users/junINOUEb/src/ens84/bioperl-live/Bio/Tree/TreeFunctionsI.pm line 94.
0 pan_troglodytes ENSPTRG00000013927 MX1-201
1 gorilla_gorilla ENSGGOG00000001560 MX1-201
2 pongo_abelii ENSPPYG00000011437 MX1-201
3 nomascus_leucogenys ENSNLEG00000008634 MX1-201
....
49 anas_platyrhynchos ENSAPLG00000015976 None
50 meleagris_gallopavo ENSMGAG00000014565 None
DBD::mysql::st execute failed: Lost connection to MySQL server during query at /Users/junINOUEb/src/ens84/ensembl/modules/Bio/EnsEMBL/DBSQL/
TranscriptAdaptor.pm line 281.
DBD::mysql::st execute failed: Lost connection to MySQL server during query at /Users/junINOUEb/src/ens84/ensembl/modules/Bio/EnsEMBL/DBSQL/
TranscriptAdaptor.pm line 281.
この場合は,以下のように set_reconnect_when_lost を行うと良いです.
my $registry = 'Bio::EnsEMBL::Registry';
$registry->load_registry_from_db(
-host => 'asiadb.ensembl.org',
-user => 'anonymous'
);
$registry->set_reconnect_when_lost;
あるいは,-host => 'asiadb.ensembl.org' に接続するだけでも問題が解消されることがあります.
|
| Genome position をもとに配列を得る |
|
slice_EnsAPI.pl.tar.gz
こちらを参照して作りました (2016 年 3 月).
[junINOUEb:Downloads]$ perl core1_db_status.pl
Slice: chromosome 12 1000000-1000050 (1)
GGATATATAGGGATAGAAGCTTGAATAGTCTGGACATATATTTGTATTGAA
|
|
一応以下のスクリプトでできます.ただし,この作業は BioMart で行ったほうがずっと早いです.
id2pos.pl.tar.gz
[inouejun:Downloads]$ perl id2pos.pl
The API version used is 83
Human, ENSG00000185960, chromosome X 624344-659411 (+1)
[inouejun:Downloads]$ perl id2pos.pl
The API version used is 67
Human, ENSG00000185960, chromosome X 585079-620146 (+1)
|
|
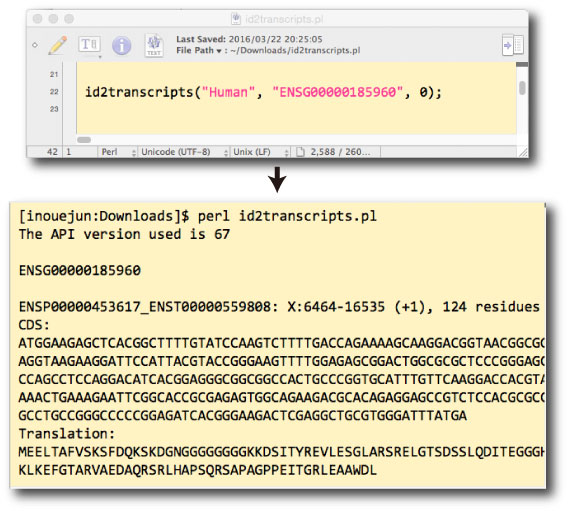
| id2transcripts.pl.tar.gz (2016 年 3 月).複数のトランスクリプトが存在する場合が多いです. |
|
|
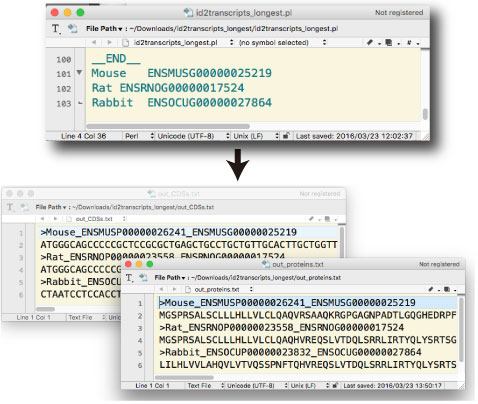
最長のトランスクリプトを得る
id2transcripts_longest.tar.gz |
|
|
| id2seq.tar.gz (2016 年 2 月), |
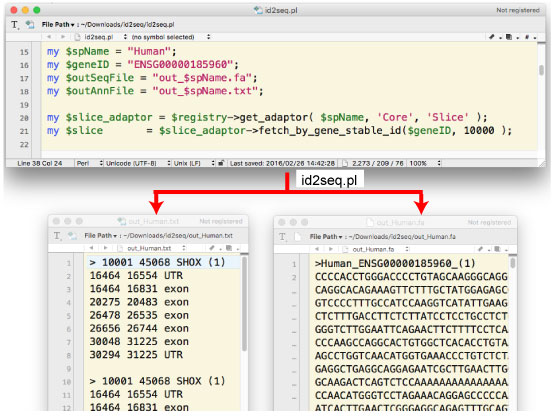 |
Ensembl API によって,種名と gene ID から,その遺伝子配列とその前後の配列を得るスクリプトを書きました.種名はこちらを参照してください.(-1) の場合は,遺伝子が reverse 鎖にコードされていることを示しています.この場合は,配列とアノテーション共に reverse and complement された結果が出力されます.
[inouejun:id2seq]$ perl id2seq.pl
UNIVERSAL->import is deprecated and will be removed in a future perl at /Users/inouejun/src/bioperl-live/Bio/Tree/TreeFunctionsI.pm line 94.
Human, ENSG00000185960, chromosome X 624344-659411 (+1)
Annotation 情報 (.txt ファイルとして出力) を取得するスクリプトも入っていますが,うまく行かないので削除しています.試す場合は,=pod と =cut の 2 つの行をコメントアウトしてください.遺伝子によって exon か UTR の start サイトを得ることができずに,以下のようにエラーが出ることが多いです.どうもsoftware と detabase のバージョンが合わないために起きる問題のようです.こちらを参照してください.
[inouejun:EnsAPI]$ perl id2seq.pl
Human, ENSG00000125798, chromosome 20 22581005-22585455 (-1)
Can't call method "start" on an undefined value at id2seq.pl line 79.
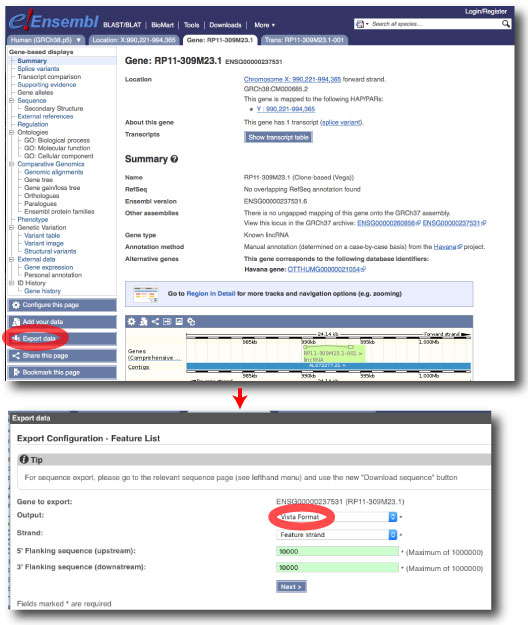
ENSG00000185960 に関する同じ情報 (VISTA format) を Ensembl Web から得る場合は,以下の手順です.fromWebExportData ディレクトリに結果を保存しました. |
|
以上,perldoc Bio::EnsEMBL::Gene (ターミナルに入力) および,Ensembl Core API Tutorial と Ensembl API を極めるを参考にしました.
|
|
| speciesName_compara.pl.tar.gz |

|
|
| classification_compara.pl.tar.gz |
|
| geneID をもとにオーソログの gene ID を得る |
|
homology_compara.pl.tar.gz (2016 年 3 月)
|
[junINOUEb:Downloads]$ perl homology_compara.pl
The API version used is 83
Query geneID: ENSG00000157601
0 pan_troglodytes ENSPTRG00000013927 MX1-201
1 gorilla_gorilla ENSGGOG00000001560 MX1-201
2 pongo_abelii ENSPPYG00000011437 MX1-201
3 nomascus_leucogenys ENSNLEG00000008634 MX1-201
4 macaca_mulatta ENSMMUG00000015329 MX1-201
5 papio_anubis ENSPANG00000023901 MX1-201
6 chlorocebus_sabaeus ENSCSAG00000008237 MX1-201
....
[junINOUEb:Downloads]$
|
エラ−メッセージ
Ensembl 83 で作成しました.fetch_by_source_stable_id() を fetch_by_stable_id() に変更する必要があるようです.Ens83 で以下のようなメッセージが出ます (2016 年 3 月).
------------------ DEPRECATED ---------------------
Deprecated method call in file homology_compara.pl line 25.
Method Bio::EnsEMBL::Compara::DBSQL::MemberAdaptor::fetch_by_source_stable_id is deprecated.
MemberAdaptor::fetch_by_source_stable_id() is deprecated and will be removed in e84. Use fetch_by_stable_id() instead
Ensembl API version = 83
---------------------------------------------------
|
|
|
|
|
|
|