|
| 2019 年 10 月 12 日 改訂 |
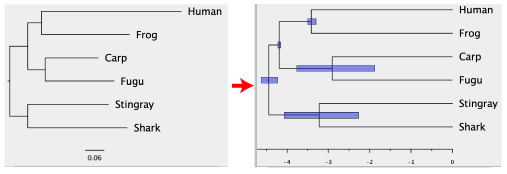 |
MCMCTREE はベイズ法に基づいて分岐年代推定を行うソフトウェアです.系統解析ソフトウェアパッケージ PAML に入っています.ここでは一般に使われている近似尤度計算 (Approximate Likelihood method) を用いた解析を説明します.
MCMCTREE で解析を行うには,Windows (XP) であればコマンドプロンプト,Mac であればターミナルを用いて,コマンドラインから操作する必要があります.このマニュアルでは,ユーザーはコマンドラインの基本的な操作ができるものと仮定して説明を行っています.
現在 MCMCTREE は頻繁にバージョンアップされているので,常にこちらから最新のバージョンをチェックする必要があります.2019 年 7 月では,バージョン 4.9i です.
このページの解析が簡単すぎる人は,mtゲノム解析編に進んでください.
English version is here.
|
| ベイズ法の利用 |
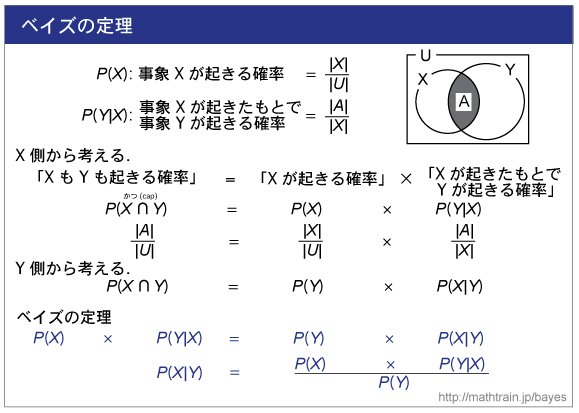 |
| こちらのサイトを参照しました. |
 |
 |
|
| MCMCTREEの特徴 |
- コマンドラインで操作.
(Windows: コマンドプロンプト; Mac: ターミナル).
- 樹形は固定.
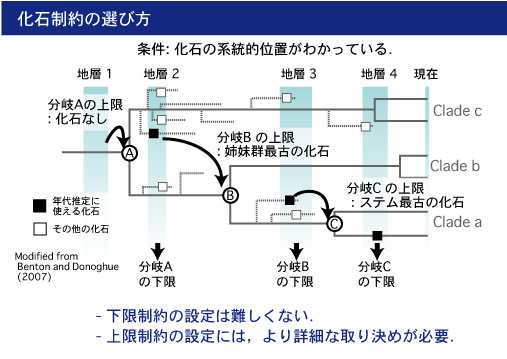
- 化石記録は事前に調査.
- 塩基,アミノ酸配列の両方で解析が可能.
- ゲノムレベルの巨大データにも対応.
- 配列データにある程度の変異が必要.
|
| 例題とソフトウェアの準備 |
例題とソフトウェアをそれぞれダウンロード & インストールしてください.ソフトウェアがダウンロードできない場合は,以下でダウンロードして得られる mcmctreeEasyExam フォルダに入っているソフトウェアを使ってください (フォルダのことをディレクトリと呼ぶこともあります).
例題
以下から例題をダウンロードしてください.
例題: mcmctreeEasyExam.tar.gz
この簡易マニュアルでは,mcmctreeEasyExam/examples フォルダにあるデータを用います.
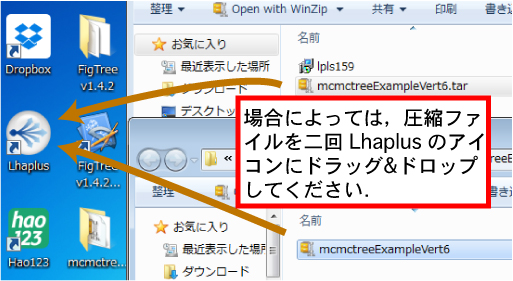
解凍プログラム [Windows のみ]
Win の場合は,.tar ファイルの解凍に Lhaplus が必要です (ただし,右クリックから .tar ファイルを解凍できる場合もあります).
- 解凍プログラム Lhaplus ( フリー) をダウンロード & インストール.
- 解凍.ダウンロードした mcmctreeEasyExam.tar.tar (あるいは mcmctreeEasyExam.tar) を,Lhaplus エイリアス (デスクトップ) にドラッグ & ドロップ.
- デスクトップに mcmctreeEasyExam フォルダが出現.
|
|
フォルダ属性の変更
[Windows]
まず拡張子 (例えば .exe) を表示します.以下のチェックを外してください.
コントロールパネル > フォルダと検索のオプション > 表示 > 登録されている拡張子は表示しない
フォルダと内部に含まれるファイルの属性を変更します. mcmctreeEasyExam/examples フォルダを右クリックし以下のチェックを外します.
プロパティ > 読み取り専用
[Mac]
カーソルで examples フォルダを選択して,コマンド+I でこのフォルダの情報を見ます.右下にある鍵マークをクリックしてパスワードを入力し,共有とアクセス権からアクセス権を「読み/書き」にします.
テキストファイルの表示
[Windows]
・試しにエディタ (メモ帳) で開いてみましょう. mcmctreeEasyExam/examples を開きます.そこに入っている baseml.ctl を右クリックして以下を選択してください.
メモ帳 で開く
(サクラエディタを用いてテキストファイルを操作しても良いです.)
[Mac]
BBEdit(Free Download が利用できます)、かあるいは mi が便利です.できれば BBEdit を使ってください.
|
解析ソフトウェアのダウンロードと準備
MCMCTREE は PAML という系統解析パッケージの一つです.PAML を http://abacus.gene.ucl.ac.uk/software/paml.html からダウンロードします.2019 年 10 月の時点では paml4.9i.tgz です. |
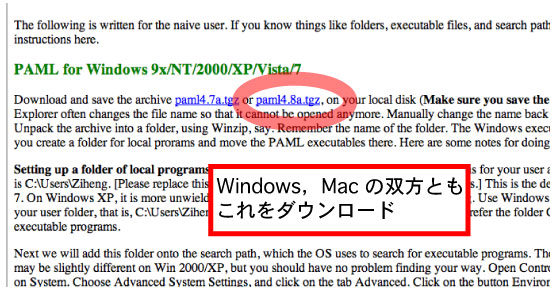 |
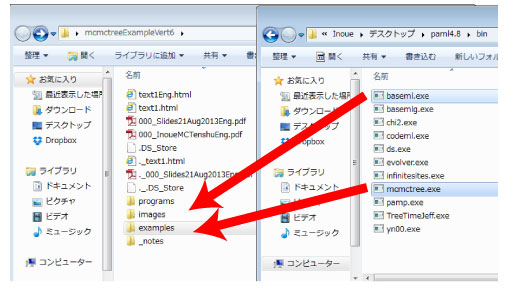
[Windows]
pamlXX/bin に入っている,
baseml.exe
mcmctree.exe
というプログラムを,以下の図を参照して mcmctreeEasyExam/examples フォルダにコピー & ペーストしてください.コンパイルせずにそのまま使えます.
|
 |
[Mac]
ダウンロードした pamlX.X.tar.gz をダブルクリックし,解凍します.terminal から解凍してできた pamlx.x/src に入ります.以下の図を参照してください. |
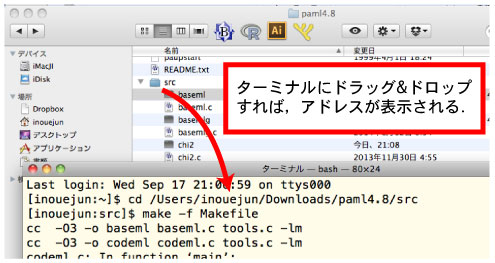 |
その後,コンパイルを行います.
make -f Makefile
src フォルダに baseml, mcmctree というプログラムが作成されます.これらを mcmctreeEasyExam/examples フォルダにコピー & ペーストしてください.
注意: 使っている Mac に make が入っていない場合は,こちらの「Xcode のインストール」を参照してください.
|
動作確認
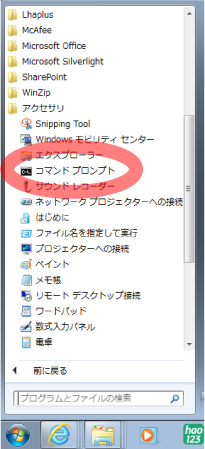
[Windows]
コマンドプロンプトから mcmctreeEasyExam/examples フォルダに入ります.コマンドプロンプトの場所は,
プログラム> アクセサリ> コマンドプロンプト
です. |
 |
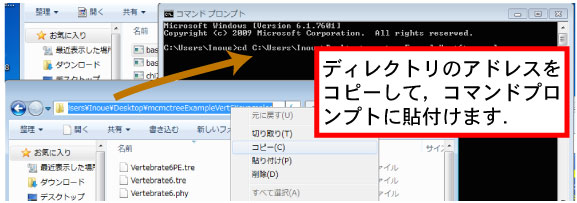
コマンドプロンプトから 「cd」 コマンドを用いて mcmctreeEasyExam/ examples フォルダに入ります.以下の図を参照して,アンダーラインで示されたアドレスをコピー&ペーストしてください.
cd xxxx/xxxx/mcmctreeEasyExam/examples
dir でフォルダに含まれるファイルを確認します.
dir
|
 |
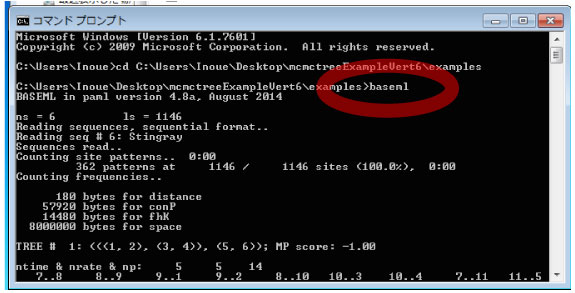
次に baseml を走らせます.コマンドプロンプトに以下のコマンドを入力してください.
(paml9i の baseml では、Substitution rate is per time unit が算出されません。この値を算出するためには、mcmctreeEasyExam/programs にある paml4.8a.tar を解凍して、古いバージョンの baseml を使ってください。)
baseml
|
 |
[Mac]
mcmctreeEasyExam/examples フォルダに入って,以下を入力してください.
./baseml
|
| インファイル |
mcmctreeEasyExam/examples フォルダに以下のファイルがあるか確認してください.上書き禁止の場合は,右クリック > プロパティで読み取り専用を解除します (Windows).
baseml.ctl
baseml を動かすためのコントロールファイル.
mcmctree.ctl
mcmctree を動かすためのコントロールファイル.
Vertebrate6.phy
シーケンスファイルです.脊椎動物 6 種の塩基配列が保存されています.
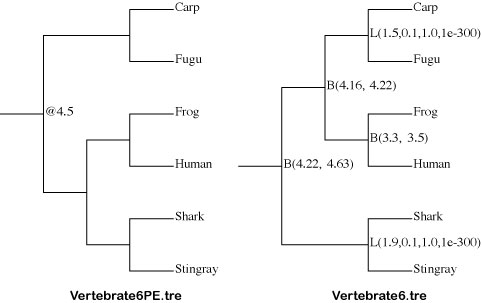
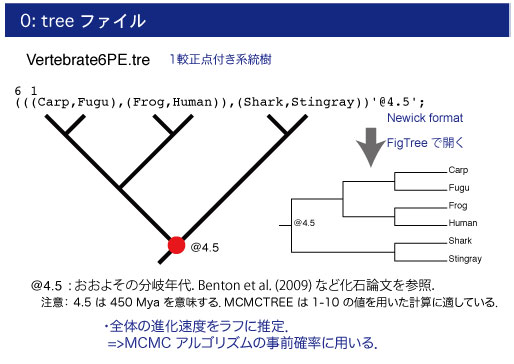
Vertebrate6PE.tre と Vertebrate6.tre
tree ファイルです.制約付きの系統樹が Newick format で保存されています.以下の操作に従って,FigTree で系統樹を確認してみましょう.
|
Tree の表示
[Windows, Mac]
FigTree をダウンロード&インストールしてください. |
|
[Windows, Mac]
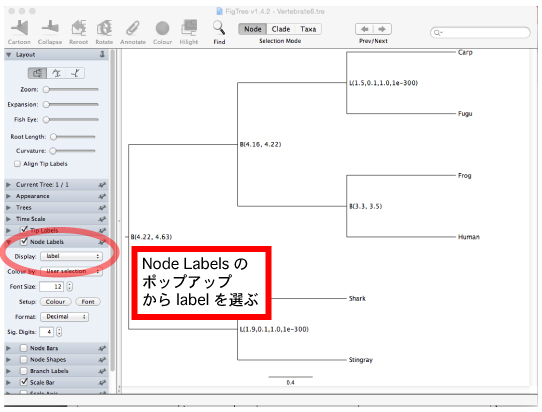
FigTree で開いて制約付き系統樹を確認します.Vertebrate6.tre ファイルを FigTree にドラッグ & ドロップ (あるいは「File > Open...」) して開きます.系統樹を以下のように表示して,分岐に与えた制約を確認してください.
バージョンによっては,自身の PC で動かないことがあります.この場合は,Java executable version をダウンロードして試してみましょう.解凍して得られた lib/figtree.jar をダブルクリックすると,FigTree が起動するはずです. Mac の場合,「figtree.jar は,開発元が未確認のため開けません.」というメッセージが出ることがあります.この場合は,「システム環境設定 > セキュリティーとプライバシー > 一般」で,「このまま開く」を選びます (figtree.jar に対するボタンが表示されるはずです)
.
|
 |
|
 |
 |
| 解析の流れ |
通常は OTU 数が多いので,近似尤度計算法 (Approsimate Likelihood Caliculation) を用いて計算を行います.近似尤度計算を用いた年代推定は,次の 2 ステップ (準備を含めると 3 ステップ) をコンピューターで別々に操作して行います.
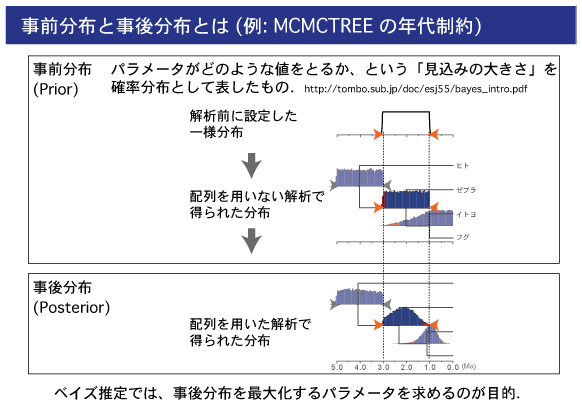
0. 進化速度のラフな推定 (MCMC 解析の事前分布として用いる)
1. 樹長の勾配ベクトルと分散共分散行列の推定 (尤度の近似に用いる)
2. MCMC アルゴリズムによる分岐年代の推定
|
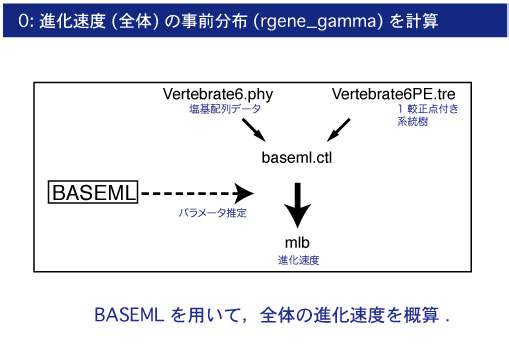
| 0.進化速度のラフな推定 |
樹長の勾配ベクトルと分散共分散行列を推定する前に,baseml を用いてデータセット全体の進化速度をラフに推定します.mcmctree.ctl ファイルに書き込む値です.paml4.9i では進化速度 (Substitution rate is per time unit) がアウトファイルに出力されません.Old versions から v4.8 の baseml を利用して算出してください (2019 年 7 月).
|
 |
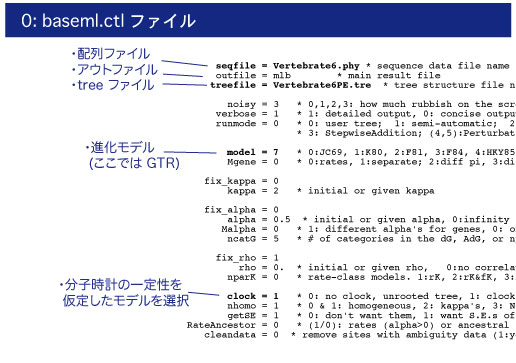
インファイルの設定
エディタで baseml.ctl ファイルを開けてください.シーケンスファイル Vertebrate6.phy と tree ファイル Vertebrate6PE.tre が読み込まれるように指定されています.GTR+G モデル (model = 7) を用いた最尤法によって進化速度を推定します.ラフな進化速度の推定のため,分子時計の一定性を仮定しています (clock = 1).詳しくはマニュアルを参照してください. |
 |
 |
 |
解析の実行
[Windows]
コマンドプロンプトから, mcmctreeEasyExam/examples フォルダに入ってください.その後,
baseml baseml.ctl
と入力して baseml を走らせます.
[
Mac]
ターミナルから,
./baseml baseml.ctl
と入力して baseml を走らせてください.以下のコマンドも同様に,「./」を入力してください.同じディレクトリにある baseml を自動的に読み込まない場合は,こちらの「.bash_profile と .bashrc」などを参照して,カレントディレクトリを読み込むように PATH の設定を行ってください.
実際には baseml.ctl を入力しなくても,baseml は自動的に baseml.ctl ファイルを読み込みます. |
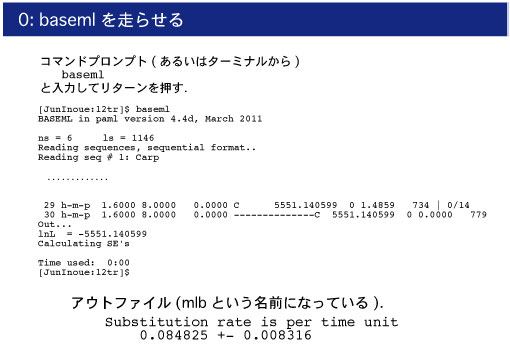 |
解析結果
結果は,mlb ファイルに保存されます.mlb ファイルをエディターで開けると,
Substitution rate is per time unit
0.084825 +- 0.008316
という単位時間あたりの進化速度が出力されています (値はバージョンによって変化しているかもしれません).この数値は,後ほど mcmctree.ctl ファイルに書き込みます.
|

|
| 1.樹長の勾配ベクトルと分散共分散行列の推定 |
| 年代推定の最初のステップでは,枝長の勾配 (gradient) とヘッセ (Hessian matrix: 枝長の分散共分散行列) を推定します.勾配とヘッセには,尤度表面の曲率を表す情報が含まれています (dos Reis and Yang, 2013, P10). |
 |
 |
インファイルの設定
エディターで mcmctree.ctl ファイルを開いてください.
usedata = 3
と書かれています.そうでない場合は,変更してください.これは,mcmctree に枝長の最尤推定値の勾配 (g) とヘッセ (H) を推定させるオプションです. |
 |
 |
解析の実行
mcmctree mcmctree.ctl
と入力します. |
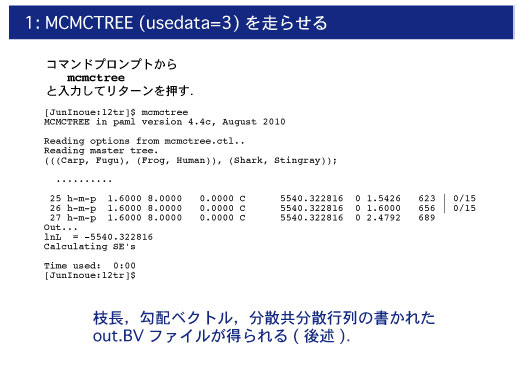 |
エラーメッセージ対策 (Mac)
「mcmctree」と入力して以下のようなエラーメッセージが出る場合があります.
[air:examples]$ ./mcmctree
MCMCTREE in paml version 4.8a, July 2014
Reading options from mcmctree.ctl..
Reading master tree.
(((Carp, Fugu), (Frog, Human)), (Shark, Stingray));
....
*** Locus 1 ***
running baseml tmp0001.ctl
sh: baseml: command not found
二つの原因が考えられます.
1) カレントディレクトリ (ここでは examples ディレクトリ) に baseml が入っていない.
2) カレントディレクトリに PATH が通っていない.
1) は単に examples ディレクトリに
baseml をコピー&ペーストして対応してください.それでも同じメッセージが出る場合は 2) を解決します.この問題は,使っている Mac でカレントディレクトリに baseml が入っているかどうか確認させる設定がなされていないために生じています.カレントディレクトリの PATH を設定するには,こちらにある「.bash_profile と .bashrc」を参照してください.
なお,.bashrc は新しくターミナルのページを開かないと作業に反映されません.以上の設定を行ったら,必ず一度ターミナルのページを閉じて,新しいページで再度 examples ディレクトリに入って解析を再開してください.
|
解析結果
推定された勾配とヘッセ行列は out.BV ファイルに保存されます.
|
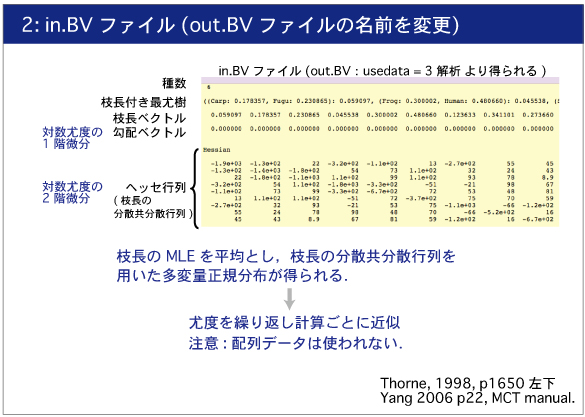
|
| 2.MCMC アルゴリズムによる分岐年代の推定 |
| ベイズ解析によって年代を推定します. |
 |
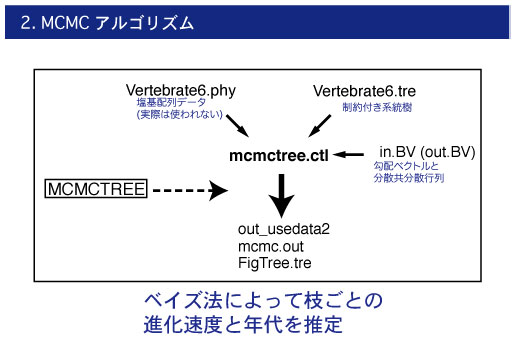 |
インファイルの設定
out.BV ファイルの名前を in.BV に変更してください.
mcmctree.ctl 開いて usedata のラインを
usedata = 2
と変更してください.このオプションによって近似尤度計算を用いた MCMC アルゴリズムを行います. アウトファイルの名前は,
outfile = out_usedata2
として変更してください. |
 |
rgene_gamma の設定
rgene_gamma は,遺伝子座の置換速度パラメーターμを設定するための事前ガンマ分布のパラメータです.事前ガンマ分布の形は,形状パラメータ (α) と 尺度パラメータ (β) によって決まります.平均を m,標準偏差を s とすると,α, β, m, s の関係は以下の式で表すことができます:
a = (m/s)^2
b = m/s^2.
例えばμの事前平均が 1 であれば,1 change/site/time unitを意味します.1 time unit が 100 MY であれば,全体の進化速度は 10^-8 substitution/site/year です.
BASEML を用いたラフな推定で得られた進化速度の値から m = 0.08 として,同様に s = 0.08 とします.すると形状パラメータ α は,
α = (0.08/0.08)^2
= 1,
となり,尺度パラメータ β は,
β = 0.08/0.08^2
=12.5
となります.このためコントロールファイルでは,
rgene_gamma = 1 12.5
と設定します.
* 複数遺伝子座の解析を行う場合 (e.g., ndata = 4) は,gamma-Dirichlet prior を設定する必要があるようです.詳細は MCMCTREE のマニュアルを参照してください (2015 年 9 月).
sigma2_gamma の設定
sigma2_gamma = 1 4.5
と設定してください. |
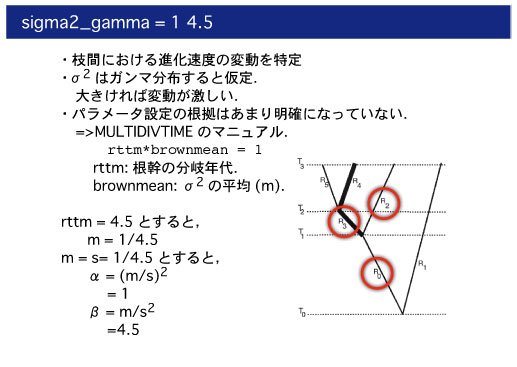 |
解析の実行
mcmctree mcmctree.ctl
MCMC アルゴリズムを走らせたらスクリーンアウトに出てくる採択率 (acceptance proportion) を見てください.これらは 30% 前後の値 (20〜40%が良いですが,15〜70%でも解析可能) が理想的です.
finetune = 1: 0.06 0.5 0.008 0.12 0.4 * times, rates, mixing, paras, RateParas
もし採択率がこの範囲内でない場合は,上記「 finetune = 1」を「 finetune = 0」にして,手動で解析を行います.この場合,Ctl+C によって解析を中断し,finetune parameters を調節して再度解析をスタートさせてパラメータを調節します. |

|
解析結果
out_usedata2 ファイルに保存されます.ここには,それぞれの分岐の推定年代と 95% 信頼区間 (CI) も書かれています.一度だけの MCMC 解析だけでは,結果は保証されません.少なくとも解析を 2 回行って,同じような結果が得られているかどうか確認してください (MCMCTREE マニュアル).
|
 |
| mcmc.txt ファイルに生データが保存されます. |
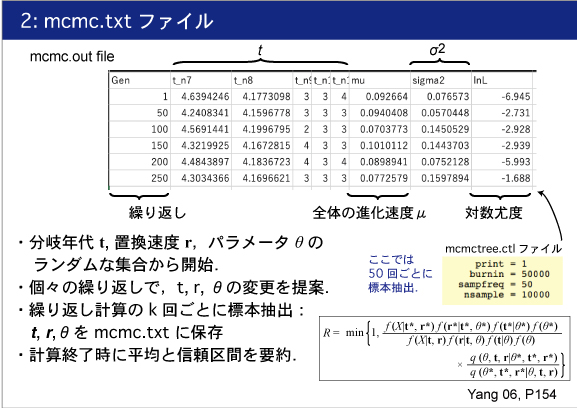 |
MCMC 出力の要約
MCMCTREE で得られる結果は,MCMC 解析で得られたサンプル (mcmc.txt に出力) をまとめて,求める年代や速度を平均値として算出しています.
真のパラメータセットの点推定として,最大事後確率を有するパラメータセットを選ぶこともできるようです (Yang 06, P167 を参照).しかし,系統樹探索の場合 (点推定 の場合,MAP 系統樹と呼ぶ) と違い,年代推定は MCMC 解析の結果得られたパラメータセット間で事後確率に大きな差異はないため,点推定よりもむしろ高い事後確率を示すパラメータ群から平均値を得て,求める年代や速度を得ているのだと思います.
事後確率の増加を目指すMCMC サンプリングを示すスライドは,こちらを参照してください (P35-55).
|
| 3.Figure を作る. |
| mcmctree によって作成された FigTree.tre ファイルを FigTree で読み込むと,以下のようなファイルが得られるはずです. |
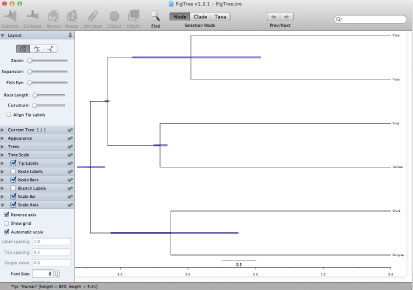
|
| Illustrator で化石制約を書き込みました. |
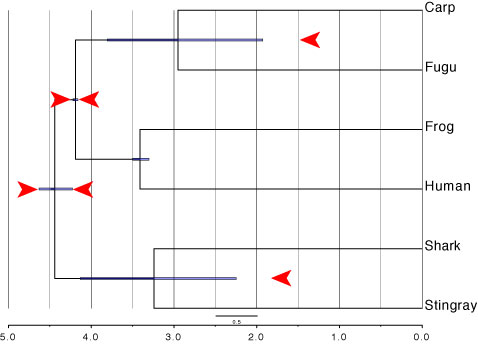
|
|
終わりです.より詳細な説明はこちらをご覧下さい.
Mega を用いて,より簡潔に年代推定を行うことも可能です.こちらを参照にしてください (2019 年 7 月).
|
参考文献
dos Reis, M., Alvarez-Carretero, S., Yang, Z. 2017. MCMCTree tutorials. PDF.
Holder, M. Bayesian Phylogenetics. PDF.
岸野洋久,Jeffrey L. Thorne. 2002. 分子進化速度のベイズ型階層モデル.統計数理.50: 17-31. PDF.
Lewis, P.O. 2011. Bayesian Phylogenetics. PDF.
Nascimento FF, Reis Md, & Yang Z. 2017. A biologist’s guide to Bayesian phylogenetic analysis. Nature Ecology & Evolution 1(10):1446-1454. PDF.
三中信宏. 2014. 系統推定論の原理と方法 -生物の系統発生を推論するロジック-.PDF.
Thorne, J. L., H. Kishino, and I. S. Painter. 1998. Estimating the rate of evolution of the rate of molecular evolution. Molecular Biology and Evolution 15:1647–1657.
Yang, Z. http://abacus.gene.ucl.ac.uk/software/paml.html
Yang , Z. 2009. 分子系統学への統計的アプローチ・計算分子進化学.藤博幸, 加藤和貴, 大安裕美,訳. 東京,共立出版. |
|
|