|
|
| 2026 年 1 月 10 日 改訂 |
 |
PAL2NAL (パルツーナル) はタンパク質コーディング領域の cDNA 配列を,アライメントされたアミノ酸配列に合わせて並べる Perl スクリプトです.cDNA 配列が第 1 座位から始まっていなくても,アライメントされたアミノ酸にあわせて,不要な部分は削除してくれます.アミノ酸配列は MAFFT や MUSCLE などであらかじめアライメントしておく必要があります.
アミノ酸ファイルと cDNA アライメントで,OTU 名が同じなくても解析可能です.順番が同じであれば動きます.ただ,両ファイルで名前の対応があっていても,同じ名前の OTU があると途中で解析がストップしてしまいます.
|
|
|
Perl スクリプトなので,コンパイルは必要ありません.
|
|
|
核遺伝子の場合
pal2nal.pl test.aln test.nuc -output fasta > outfile
test.aln : アライメント済みファスタ形式のアミノ酸配列.
test.nuc: ファスタ形式の塩基配列.
-output fasta: outfile をファスタ形式で保存.
注意:アミノ酸と塩基配列のファイルは,OTU 名が一致していれば順番が同じである必要はありません.順番が同じであれば,OTU 名前が異なっていても解析可能です.
PAL2NAL_Example1.tar.gz |
 |
ミトコンドリア遺伝子の場合 [うまく動きませんでした]
pal2nal.pl test.aln test.nuc Nuc -codontable vmitochondria -output fasta > Outfile
-codontable vmitochondria: Genetic code をミトコンドリアにします.
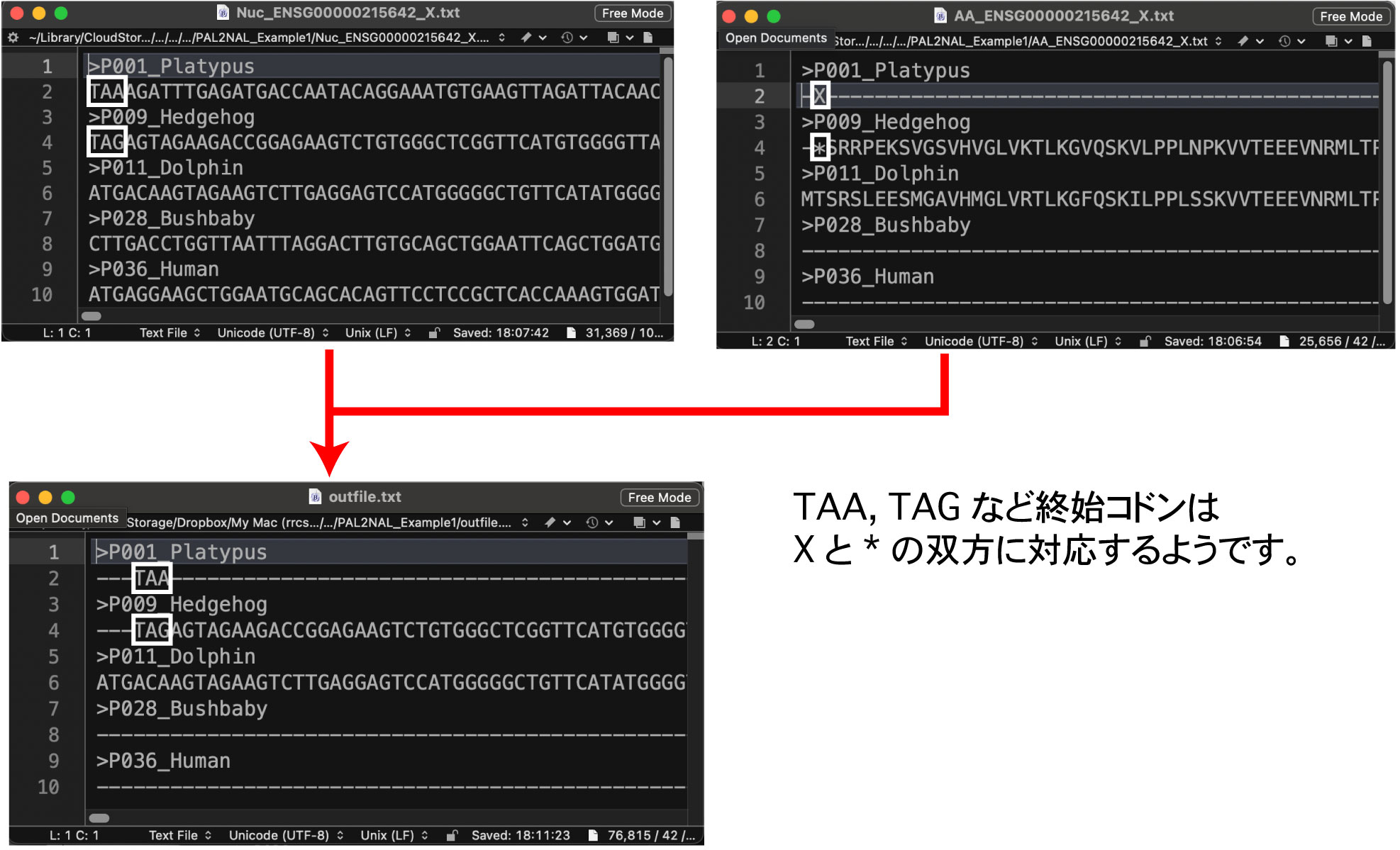
* 終止コドンが不完全な場合は,塩基配列が 3 の倍数で終わっていないことがあります.この場合はアウトファイルが空で出てくるので,注意してください.
|
|
|
- 塩基配列の開始コドンより前に余分な配列 (UTRs: Untranslated Regions) がある場合は,塩基配列のアライメントでは削除されています.たとえばこちらの例題では,開始コドン前の UTRs 1806 bp が削除されて,塩基配列のアライメントがアミノ酸アライメントに合わせて作成されています.
このことは PAL2NAL の website にも書いてあります."The program automatically assigns the corresponding codon sequence even if the input DNA sequence has mismatches with the input protein sequence, or contains UTRs, polyA tails. "
- ギャプだけからなる OTU があると,アライメントが動かないようです.
- 塩基配列の長さがコドンで終わっていないで,1,2 塩基余っていると,エラーメッセージとともに空のファイルが作成されるようです.ミトコンドリアゲノムにコードされているタンパク質遺伝子は終止コドンが不完全な場合があるので,注意する必要があります.
|
|
PAL2NAL で以下のようなエラーメッセージが出ることがあります.
#--- ERROR: inconsistency between the following pep and nuc seqs ---#
>ANg10010t1
MDEIMLVNRNDTF.....
>ANg10010t1
ATGGATGAAATCA.....
Run bl2seq (-p tblastn) or GeneWise to see the inconsistency.
この場合は,こちら (BLAST Basic Local Alignment Search Tool をクリック) から local Blast (Blast+ ではありません) をダウンロードしたときに得られる bl2seq で解析すると,cDNA とアミノ酸配列の相違点を表示してくれます.私は blast-2.2.26-universal-macosx.tar.gz をダウンロードしました.
./bl2seq -p tblastn -i pep.seq -j nuc.seq -F F
pep.seq
エラーメッセージで指摘されたアミノ酸配列.
nuc.seq
エラーメッセージで指摘された cDNA 配列.
詳細は bl2seq_p2n.tar.gz をご覧下さい.
|

|
| bl2seq の代替方法: 2 つの配列を比較する |
|
NCBI の bl2seq ツールは、古い「legacy BLAST」パッケージの一部で、現在の BLAST+ には含まれていません。代わりに、BLAST+ のコマンド(blastn, blastp, tblastn など)でペアワイズ比較が可能です。
tblastn_p2n.zip
2026 年 1 月
|
|
こちらにある「cDNA アライメントのフレームシフトチェック」を参照してください.
cDNA_AA_comparison.tar.gz
(2018 年 3 月)
|
| Perl script: 複数のファイルを PAL2NAL でアライメントする |
|
#!/usr/bin/perl -w
strict;
use warnings;
my $job = '';
my @files = ();
opendir (DIR,".") || die "cannot open :$!\n";
@files = readdir(DIR);
close(DIR);
foreach my $nameline (@files) {
if ($nameline =~ /^AA\_/) {
$nameline =~ /AA\_(\w+)/;
$job = $1;
print "$job\n";
system("pal2nal.pl AA\_$job Nuc\_$job -output fasta > outNuc\_$job");
print "Done $job\n";
}
}
print "Done all alignments\n";
exit;
例題はこちらです.
|
|
|