|
|
|
|
系統解析に使えそうな Perl script をつくっています.簡単な参考書やこちらのサイトを参照しながら使ってください.
|
|
通常 Mac には perl がインストールされています.以下のコマンドによって,バージョンを確認してください.
perl -v
私はアップグレードしたかったのですが,やり方がわからず (アップグレードは無いのかもしれません),結局インストールしました.こちらから最新版をダウンロードしてください (The latest releases in each branch という表です).ただし,5.13 などのように奇数は安定していないようなので,偶数を選んだ方が無難です.私は 5.12.2 をダウンロードしました.解凍して得られたフォルダに入り,以下の順番でコマンドをうちます.全体で 10 分位かかったでしょうか.
$ sh Configure -de
$ make
$ sudo make install
$ make clean
make test もやるように指示されますが,どちらでも良いようです.make test だけでも 5 分位かかったような気がします.
|
|
以下を test.pl として保存します.ターミナルから,「perl test.pl」と入力してください.
#!/usr/bin/perl
use strict;
use warnings;
print "Hello World.\n";
|
|
|
|
塩基配列以外の文字を取り除く
$line =~ s/[^a-z]//g;
ギャップを取り除く
$sequence =~ s/-//g;
|
|
複数ファイル内部にある特定の文字を変更するには, こちらのページを参照して下さい.
例 1) html ファイルの内部を変更する.
perl -i.bak -p -e 's/#ffffdd/#aaaacc/ig;' *.html
拡張子が .html になっているファイル内部を検索し,#ffffdd を #aaaacc に変換します.'s/\n/\r/ig;' にすれば,改行コードを Unix から Mac に変換します.
-i に .bak をつけると,バックアップファイルを作ってくれます.
-i は上書き保存です.-i (ここでは -i.bak) をはずして,最後に > outfile をつけると,上書きではなくなりますが,ワイルドカード (*) を用いて複数のファイルを一気に変換できなくなります.
|
|
system および opendir 関数を使って,他の perl script を動かします.
perl PerlController.pl PerlScript.pl
PerlController.pl: 対象となる perl script.操作する script は INFILE と OUTFILE を画面に入力するスクリプトである必要があります (例: perl PerlScript.pl infile.fas > outfile).
PerlScript.pl: p distance を計算するプログラムです.PAUP や PAML など他のプログラムと比較して,注意して使ってください.
PerlContoroller.pl
|
 |
|
主に以下のテクニックを使っています.
アウトプットディレクトリを作成する.
my $OutFolder = "Out_fol";
# Output folder is made.
if (-d "$OutFolder") {
system ("rm -r $OutFolder");
}
if (!-d "$OutFolder") {
mkdir("$OutFolder", 0755);
}
ディレクトリに入っているファイル名を配列に格納する
opendir (DIR,".") || die "cannot open :$!\n";
@dirfile = readdir(DIR);
close(DIR);
System 関数によって他のプログラムを操作する.
system("perl PERLSCRIPT.pl INFILE > OUTFILE");
|
|
|
|
|
|
|
#!/usr/bin/perl
use strict;
use warnings;
use Cwd;
my $cur_dir = getcwd();
print "Current directory:\n$cur_dir\n";
|
|
|
Unix から Mac 形式に改行コードを変換します.
LineBreakChange.tar.gz
参考になるサイト.
http://osksn2.hep.sci.osaka-u.ac.jp/~taku/osx/crlf.html
|
| grep を使ってアレイ内部に文字列があるか調べる |
|
@array = ("apple", "application","pineapple","wine","windows");
$count = grep(/^app/, @array);
@items = grep(/^app/, @array);
print "件数は $count、内容は@items.\n";
実行結果は以下の通り.
件数は2、内容はapple application.
* 完全一致.
if(grep($_ eq "apple", @array))
* メタ文字を文字そのものとして判断させるには m|\Q〜\E| を使う.
if(grep m|\Qhttp://www.geocities.jp\E|, @array)
補足. メタ文字を文字そのものとして判断させる手法は,かなり便利.
$genomesHtmlLine =~ s|\QLoss(0)<br>= XXX</td>\E||g;
|
|
|
my $count = $line =~ tr/,//;
print "$count\n";
|
|
|
「out_ENSG00000000003_ENST00000373020」
-> 「ENSG00000000003_ENST00000373020」
NameChange_fol.tar.gz
|
|
matchCounter.pl.tar.gz (2016 年 3 月)
こちらを参照しました. |

|
|
fastaReadHash.tar.gz (2017 年 12 月)
|
|
#!/usr/bin/perl -w
# perl fasHash.pl db.fas > out.fas
use strict;
use warnings;
my $infile = "db.fas";
open(INFILE,"$infile") or die "$!";
my @inFileCont = ;
close INFILE;
my ($ref_fasHash,$ref_nameArray, $sequenceLength)
= &fastaReadHash(\@inFileCont);
foreach my $name (@$ref_nameArray){
print "$name\n";
print "$$ref_fasHash{$name}\n";
}
exit;
sub fastaReadHash
{
my $name = "";
my $sequence = "";
my $sequenceLength = "";
my $flag = 0;
my @nameArraySR = ( );
my %fasHashSR = ( );
my ($inFileContSR) = @_;
foreach my $inFileLine (@$inFileContSR) {
chomp($inFileLine);
if($inFileLine =~ /^>/){
if($flag == 0){$name = $inFileLine;
$flag = 1;
} else {
$fasHashSR{$name} = $sequence;
push(@nameArraySR,$name);
$name = "$inFileLine";
$sequence = "";
}
} else {
$sequence .= $inFileLine;
}
}
$fasHashSR{$name} = $sequence;
$sequenceLength = length($sequence);
push(@nameArraySR,$name);
return (\%fasHashSR,\@nameArraySR,\$sequenceLength);
}
|
| 大規模データベースから keyword を含むレコードを取り出す |
|
 |
fastaRecPickUpNoMemory.tar.gz (2017 年 12 月)
|
| Fasta 形式のファイルを二次元配列に読み込むサブルーチン |
|
| fasToPhy2D.tar.gz |
#!/usr/bin/perl -w
use strict;
use warnings;
my $inFile = "infile.txt";
open(IN,"$inFile") or die "Cannot find $inFile\n";
my @inFileCont = <IN>; close IN;
my ($refRec, $totalLength) = &fasReader2d(\@inFileCont);
my @recs = @$refRec;
foreach my $ele (@recs) {
print "$ele->[0]\n";
my @tempSeq = @{$ele->[1]};
foreach my $ch (@tempSeq){
print "$ch"; } print "\n";
}
exit;
sub fasReader2d{
my $uniqNameSR = "";
my $seqLength = "";
my $flag = 0;
my @recs2D = ();
my @tempSeq = ();
my $tempSeq = "";
my ($inFileContSR) = @_;
foreach my $inFileLine (@$inFileContSR) {
chomp($inFileLine);
if($inFileLine =~ /^>/){
if($flag == 0){
$uniqNameSR = $inFileLine;
$flag = 1;
}
else {
my @tempSeqArray = split(//, $tempSeq);
print "tempSeq $tempSeq\n";
push(@recs2D, [$uniqNameSR, \@tempSeqArray]);
$uniqNameSR = $inFileLine; $tempSeq = "";
}
}
else {
$tempSeq .= $inFileLine;
}
}
my @tempSeqArray = split(//, $tempSeq);
push(@recs2D, [$uniqNameSR, \@tempSeqArray]);
$seqLength = scalar(@tempSeq);
return (\@recs2D, $seqLength);
}
|
|
| revCom.tar.gz |

|
sub revCom {
my ($seqLine) = @_;
my $revcomp = reverse($seqLine);
$revcomp =~ tr/ATGCatgc
/TACGtacg/;
$revcomp =~ tr/atgc/ATGC/;
return ($revcomp);
}
|
| Fasta: 配列を長い (短い) 順に並び替える |
|
| lengthOrder.tar.gz (2013 年 6 月) |

|
| Fasta: Database からキーワードを含むレコードを抽出する |
|
キーワードが name line に含まれるレコードをデータベースから抽出します.
retRecords.tar.gz (2016 年 7 月)
|

|
|
fasta 形式のファイルから配列を切り出します (2017 年 7 月).
% perl SeqSlicer.pl seqFile.txt scaffold_66 10 20
aggtgtgctt
SeqSlicer_fol.tar.gz
|
|
|
|
infile 内部にある OTU 名の変換を行い,outfile に書き出します.
「AB005035」-> 「Lancelet_AB005035」
list に返還前の名前と返還後の名前をリストアップしておきます.
FasNameChange_fol.tar.gz
複数のファイル内の文字列を変更する場合は,こちらが参考になります.
|
|
|
リストアップしたOTU 名に従って sequence file を並び替えます.fasta file にリストアップした OTU 名がない場合は,短いギャップ配列が書かれます.リストアップした OTU 名前と sequence file の OTU 名は一致させてください [2010 年 12 月].
FastaOrderChanger_fol.tar.gz
|
|
|
|
|
|
|
全ての種名をリストアップしたファイルを作成してください.このリストとファスタファイルの種名は完全に一致している必要があります.配列が無い遺伝子は,他の種と同じ長さのギャップが加えられます.大規模データへの適用を考えて,メモリーを使わないようにインファイルから 1 種づつ配列を取り出してアウトファイルに書き出します.配列はアライメントされている必要があります [2010 年 12 月].
SeqConcatBD_fol.tar.gz
|
|
|
| fasToPhy.tar.gz (2016 年 3 月). |

|
| Fasta: match first に置き換える |
|
| fasToMF.tar.gz [2016 年 3 月] |

|
|
| fasToMF_return.tar.gz [2016 年 7 月] |

|
| Fasta: phylip 形式に Site 番号をつける |
|
| fasToPhy_num.tar.gz (2016 年 3 月) |

|
|
motifFinder.tar.gz
$match に入力したモチーフを探します.
my $match = "TCACA[CG]";
TCACACG か TCACACC を表します.
my $match = "T..CACCT";
.. に任意の 2 文字が入っていることを意味します.
(2016 年 3 月) |

|
|
| motifFinder_region.tar.gz |

|
| Fasta: cDNA 配列を色付き html に書き換える |
|
アライメント済み Fasta 形式の cDNA を,コドン別に色分けし,match first にして見やすくしたアライメントファイル (html) に変換するプログラムです.
fasToPhyInt.tar.gz [2017 年 1 月]. |

|
| Nexus: cDNA 配列を色付き html に書き換える |
|
Mesquite で保存した cDNA nexus フォーマットを以下のように色付き html ファイルに変換します.イントロン部分は色を付けず下線にします.
nex2PhyInt.tar.gz
|

|
|
Coding sequence (DNA) を 1st, 2nd, 3rd からアミノ酸配列に翻訳します.
perl cDNATranslater.pl > out.txt
cDNA_AA_comparison.tar.gz (2019 年 1 月)
|

|
|
my @nameArrays = (Xenopus8, Cod4, Fugu5)
my $longestName = &logestNamePicker(\@nameArrays);
sub logestNamePicker {
my ($refNames) = @_;
my @nameArrays = @$refNames;
my @nameArrays2D = ( );
foreach my $nameSR (@nameArrays){
push(@nameArrays2D,[length($nameSR), $nameSR]);
}
my @name2dSorted = sort{$b->[0] <=> $a->[0]}@nameArrays2D;
my $longestName = $name2dSorted[0][1];
return($longestName);
}
|
|
|
得られたファイルを MacClade で開くと,アミノ酸翻訳に対応した DNA アライメントを見ることができます.
FasToNex_DNA.pl.tar.gz: 塩基配列用です [2010 年 11 月].
FasToNex_AA.pl.tar.gz: アミノ酸配列用です [2010 年 11 月].
FasToNex_cDNA.pl.tar.gz: cDNA 配列用です.インファイルを「.fas$」とすることで,.fas で終わるファイルをすべて変換します.遺伝暗号は NCBI にあるこちらのサイトを参照しました. [2010 年 12 月].
|
|
|
|
|
|
FasDelIdenSeq_fol.tar.gz
|
|
|
|
|
| Fasta: タンパク質転写領域をコドン別に分ける |
|
| CodonSeparatorNew.tar.gz |

|
| Fasta: コドン別の配列 -> cDNA 配列 |
|
|
| Fasta: タンパク質転写領域の 3rd position を削除する |
|
3rdDeleter_fol.tar.gz
|
|
| NucTranslaterP2N.tar.gz (2017 年 11 月) |
 |
pal2nal に genetic code など設定をできるだけあわせています.例えば,TNC (N が含まれている) や A-A (ギャップが含まれている) などのコドンは X として返します.MacClade ではこれらのコドンを,アミノ酸翻訳ではギャップとして返しているので,相違が見られます.
ミトコンドリアの遺伝子暗号も付け加えました (2017 年 11 月).
|
| mRNA 配列から 5' 末端の非翻訳領域 (UTRs) を取り除く |
|
| CDSfinder.tar.gz (2014 年 12 月) |

|
| トランスクリプトームからタンパク質遺伝子配列を抜き出す |
|
 |
[inouejun:tBlastNResPicker]$ perl 010_autoQueryMaker.pl
1.ENSP00000375041
2.ENSP00000374824
3.ENSP00000374825
[inouejun:tBlastNResPicker]$ cd 020_transcripts/
[inouejun:020_transcripts]$ sh command.sh
Building a new DB, current time: 01/23/2015 19:12:29
New DB name: transcripts.fas
New DB title: transcripts.fas
....
[inouejun:020_transcripts]$ cd ../
[inouejun:tBlastNResPicker]$ perl 030_autoTblastn.pl
1.ENSP00000374824.txt
2.ENSP00000374825.txt
3.ENSP00000375041.txt
[inouejun:tBlastNResPicker]$ perl 040_autoSixWayTranslation.pl
#### 1.ENSP00000374824.txt ####
#### 2.ENSP00000374825.txt ####
#### 3.ENSP00000375041.txt ####
[inouejun:tBlastNResPicker]$ |
tBlastNResPicker.tar.gz (27MB)
|
| Fasta: アミノ酸ファイルに合わせて cDNA をアライメント |
|
アミノ酸アライメントに合わせて,cDNA のアライメントを作成するスクリプトです [2017 年 12 月 改訂].
./gapAddCDNA.pl AAincGap.fas DNAnoGap.fas
AAincGap.fas: アライメント済みのアミノ酸配列
DNAnoGap.fas: ギャップなしの cDNA 配列.
二つのインファイルで OTU 名は一致している必要はありませんが,順番は一致させてください.アミノ酸と cDNA トリプレットの対応はチェックせずに,純粋にギャップの位置だけでアライメントを作成しています.
インファイルのギャップを取り除いた状態でアミノ酸と DNA で長さを比較してスクリーンアウトしていますが,あまり必要ないような気がしてきました.長さが異なることがよくありますが,これは「A-T」などはアミノ酸配列では X などになっているためだと思われます.
UTRs やポリ A が入っている場合は,一度 pal2nal で処理してこれらを取り除く必要があります.
gapAddCDNA_fol.tar.gz
|
|
DNA を相補鎖に変換するプログラムです.ヒントだけ書きます.詳しくはこちらを参照してください [2015 年 1 月].
# reverse the DNA sequence
my $revcomp = reverse($dna);
# complement the reversed DNA sequence
$revcomp =~ tr/ABCDGHMNRSTUVWXYabcdghmnrstuvwxy
/TVGHCDKNYSAABWXRtvghcdknysaabwxr/;
revCom.pl.tar.gz
|
|
重複したレコードを削除するスクリプトです.ハッシュを使っていないので,レコードの順番を変更しません [2011 年 5 月].
uniqSeqPicker_fol.tar.gz
|
sub uniqArrayOneslf{
my $refArray = shift@_;
my @uniqArray;
foreach my $name (@$refArray) {
if(grep($_ eq "$name", @uniqArray) == 0){
push(@uniqArray,$name);
}
}
return \@uniqArray;
}
sub printSequence {
my($seqSR, $lengthSR) = @_;
for (my $pos = 0 ; $pos < length($seqSR) ; $pos += $lengthSR ) {
print substr($seqSR, $pos, $lengthSR), "\n";
}
}
|
| Phylip 形式の配列を Fasta 形式に書き換える |
|
| phySeq2fas.tar.gz (2016 年 3 月) |

|
| Phylip 形式の tree ファイルを nexus 形式に変換 |
|
perl TreePhyNex.pl infile.PHYLIP > outfile.nex
TreePhyNex.pl.txt
|
|
NexToFas_fol.tar.gz [2010 年 12 月]
サブルーチンの抜粋は以下
my($ref_seqHash,$ref_nameArray) = &nexReader(\@inFileCont);
my %seqHash2 = %$ref_seqHash;
my @nameArray2 = @$ref_nameArray;
# プリントアウト
open(OUT,">$outFile");
foreach (@nameArray2) {
print OUT ">$_\n$seqHash2{$_}\n";
}
###### サブルーチン
sub nexReader
{
my $stone = 0;
my @nameArray = ( );
my %seqHash = ( );
my ($ref_inFileCont) = @_;
my @inFileCont = @$ref_inFileCont;
foreach my $line (@inFileCont) {
chomp($line);
if($line =~ /^END;/){ # 解析終了.
$stone = 0;
}
if($stone == 1) { # MATRIX〜END; 間の解析
if($line =~ /^[a-zA-Z0-1]/) { # sequence のある行を認識
$line =~ s/ +\[\d+\]//g; # スペース[数字] を除去
my($name,$seq) = split(/ +/,$line);
# @nameArray に OTU 名があれば
#ハッシュだけ作って OTU 名を新たに格納しない.
if(grep($_ eq "$name", @nameArray)){
$seqHash{$name} .= $seq;
next;
# @nameArray に OTU 名があれば,OTU 名を格納し,
# ハッシュを作成する.これは最初の段だけ.
} else {
push(@nameArray,$name);
$seqHash{$name} .= $seq;
}
}
}
if($line =~ /^MATRIX/){ # 解析開始.
$stone = 1;
}
}
return(\%seqHash,\@nameArray);
}
|
| Nexus 形式を phylip 形式に書き換える |
|
これもNexus 形式の配列部分に,改行を含めないでください.Match first にも対応していないです.
NexToPhy_fol.tar.gz
|
| Nexus 形式: MacClade でマークしたサイトを取り除く*** |
|
MacClade のコメント欄に「#」と記入したサイトを取り除きます.結果をFasta 形式で保存します.MacClade で保存したファイルの改行コードは Mac 形式ですが,自動的に Unix 形式に変換します.
NexToPhy_fol.tar.gz
|
| GenBank 形式: アミノ酸配列と塩基配列を抽出する |
|
複数の種から得られた相同遺伝子の cDNA 配列が保存された GenBank format (一つにつながったファイル) から,アミノ酸配列と塩基配列を抽出してアライメントを行います.GenBank format は何種入っていても大丈夫ですが,遺伝子は 1 つだけです.遺伝暗号の関係上,ミトコンドリア遺伝子には対応していません (pal2nal はミトコンドリア遺伝子では正常に作動しないためです) [2010 年 10 月].
GenBankParser_AA_DNA.pl sequence.gb
上記のコマンドによって,mafft (インストールする必要があります),pal2nal, FasToNex_cDNA.pl が自動的に作動して,アミノ酸のアライメントが行われ,cDNA のアライメントも得られます.
GenBankParser_AA_DNA_fol.tar.gz
|
| GenBank 形式: mt ゲノムファイルからアミノ酸配列を抽出する |
|
mt ゲノム配列が保存された GenBank format (複数の種) からアミノ酸配列を抽出するスクリプトです.塩基配列は同じ GenBank format を GenBankStrip.pl によって処理すれば得られます.OTU 名にスペースが入っていると MacClade で読み込むときなどに問題になるので,Editor を使って変更する必要があります [2010 年 10 月].
aaSeqRetFromGBK_fol.tar.gz
|
|
|
P distance (変異サイト/全長) を計算します.paup より若干低い値が出ます.理由は調べているところです.
Pdistance.pl
E001 vs E001 Seq Lenght: 789; Dif sites: 0; P dist: 0.000
E001 vs E002 Seq Lenght: 789; Dif sites: 50; P dist: 0.063
E001 vs E004 Seq Lenght: 789; Dif sites: 58; P dist: 0.074
E002 vs E001 Seq Lenght: 789; Dif sites: 50; P dist: 0.063
E002 vs E002 Seq Lenght: 789; Dif sites: 0; P dist: 0.000
E002 vs E004 Seq Lenght: 789; Dif sites: 45; P dist: 0.057
E004 vs E001 Seq Lenght: 789; Dif sites: 58; P dist: 0.074
E004 vs E002 Seq Lenght: 789; Dif sites: 45; P dist: 0.057
E004 vs E004 Seq Lenght: 789; Dif sites: 0; P dist: 0.000
PAUP
E002 Dolphin E001 Human 0.06802721
E004 Megabat E001 Human 0.07900394
E004 Megabat E002 Dolphin 0.06131519
PdistAdv_fol.tar.gz
* 小数点以下 3 桁まで表示します.
$formated = sprintf( "%.3f", $pdist);
|
| Newick tree: 系統樹の順番に配列を並べ替える |
|
jason2seqnece.tar.gz
解析に際し,FigTree などを使って,newick format を Jason format に書き換える必要があります (2018 年 12 月).
|

|
|
|
sequence file に並べられた順番を番号として,tree file の種名を番号に置き換えます.
SpNameToNum_fol.tar.gz
|
| Newick tree: クレードごとに OTU を書き出す |
|
my $tree = '(4,(3,(2,1)))';
my @content;
while ($tree =~ s/\(([^\(\)]+)\)/$1/) {
my $clade = $1;
my @temp = split(/,/, $clade);
push(@content, \@temp);
}
for (my $i = 0; $i < scalar(@content); $i ++) {
print("content of clade $i: " . join(', ', @{$content[$i]}) . "\n");
}
二分探索木 (binary search tree) |
| Newick tree: 大きい順にクレードを書き出す |
|
newick 形式を読んで,クレードに含まれる leaf をクレードの大きい順に書き出す.
cladePicker.pl.tar.gz
|
|
二つの系統樹に含まれる OTU 構成は同じにしてください.遺伝子の系統樹 (比べる対象) の OTU 名の最初は,種の系統樹で用いた OTU 名にして,「_」(アンダーバー) を挟んで遺伝子名などを書くようにしてください [2012 年 3 月].
treeComparison.tar.gz |
|
[inouejun:treeComparison]$ perl treeComparison.pl
Number of reconstructed nodes: 3
6 sp clade
4 sp clade
2 sp clade |
|

|
| Newick tree: ヒトと真骨類が含まれるクレードを抜き出す |
|
Human (_H)と teleosts ('_Z','_M','_S','_T') の両方が含まれる最小のクレードを選んで (下図),leaf を書き出します.
Notung (NHX) format
用:cladePickerNTform_fol.tar.gz (不完全)
Newick format 用:multiCladePickerNewick_fol.tar.gz (おすすめ)
[inouejun:multiCladePickerNewick_fol]$ perl multiCladePickerNewick.pl
ADCY1_H: ADCY1_C ADCY1_H ADCY1_X ADCY1a_G ADCY1a_T ADCY1a_M ADCY1b_G ADCY1b_T ADCY1b_M
NonFishClades
9, ADCY1_C ADCY1_H ADCY1_X ADCY1a_G ADCY1a_T ADCY1a_M ADCY1b_G ADCY1b_T ADCY1b_M
3, ADCY1_C ADCY1_H ADCY1_X
2, ADCY1_C ADCY1_H
FishClades
6, ADCY1a_G ADCY1a_T ADCY1a_M ADCY1b_G ADCY1b_T ADCY1b_M
3, ADCY1a_G ADCY1a_T ADCY1a_M
3, ADCY1b_G ADCY1b_T ADCY1b_M
2, ADCY1a_G ADCY1a_T
2, ADCY1b_G ADCY1b_T
ADCY8_H: ADCY8_2_M ADCY8_2_T ADCY8_C ADCY8_H
NonFishClades
4, ADCY8_2_M ADCY8_2_T ADCY8_C ADCY8_H
2, ADCY8_C ADCY8_H
FishClades
2, ADCY8_2_M ADCY8_2_T
(以下は問題があるので使わないでください)
Human-Teleosts クレードに含まれるクレードのうち,teleosts だけからなる全クレード (下図) をプリントアウトするスクリプトを書きました.
Notung (NHX) format
用:cladeChoserNTform_fol.tar.gz
Newick format 用:cladeChoserNewick_fol.tar.gz
[inouejun:cladeChoserNTform_fol]$ perl cladeChoserNTform.pl
Fish clades within Human-Teleostei clade
n31:0.007932714818834855[&&NHX:S=TetStic:D=N:B=70.0]
Stickleback_ENSGACP00000010656 Tetraodon_ENSTNIP00000007126
r101[&&NHX:S=Percomorpha:D=N]
Stickleback_ENSGACP00000010656 Tetraodon_ENSTNIP00000007126 Medaka_ENSORLP00000017625
n35:0.02428732280764367[&&NHX:S=Clupeocephala:D=N:B=70.0]
Stickleback_ENSGACP00000010656 Tetraodon_ENSTNIP00000007126 Medaka_ENSORLP00000017625 Zebrafish_ENSDARP00000021652
|

|
|
外群 1 種の樹長付き rooted tree を解析して,根幹から末端までの枝長を種ごとに計算します [2011 年 11 月].
blCalculator.tar.gz
|
[inouejun:BranchLengthR]$ perl blCalculator.pl
outgroup: SeaSquirt
Blanch length from root to top:
1.3: Stickleback
0.7: Fugu
0.5: Medaka
0.4: Zebrafish
0.7: Chicken
0.6: Human
0.4: Xenopus
0.2: SeaSquirt
|

|
|
思うような結果が得られませんでしたが,日本語版 Yang (2006) P 217 にある Fitch (1976) の方法で相対速度テストを行ったので解説します.例として,上の図にある枝長付き系統樹を使っています.他にも相対速度テストが紹介されていますが,標準誤差を配列を解析して求めたりする必要があり,perl だけで簡単にできそうにないので試していません.
ここでは Human と他種の間で枝長を比較し,カイ二乗適合度検定によって他種の枝長が棄却できるか (あり得ない長さか) を検定しています.Yang (2006) では 5 行目における枝長の推定が,BL を 2 倍したものになってしまっているようにも受け取れるので,BL と BL*2 の両方を計算しました.この本によると,カイ二乗適合度検定は自由度 1 で行うようです.
上の図のように,本来ならば Stback が棄却されてほしかったのですが,カイ二乗 (X^2) の値は,BL2 であっても 0.5157.. と,有意水準 0.1 (P=0.1) の値 2.706 (カイ二乗分布表より) に遠く及ばないです. 試しに BL を 2.8 にすると,ようやく BL2 で計算した場合は P=0.1 で棄却できました.この場合,Human の約 4.7 倍にもなってしまいます.この方法がおかしいのか,私の解釈が間違っているのか,今のところ解決できていません.実際のデータを使っても同じような結果が得られてしまいました [2011 年 11 月].
=>平均値 (枝長間) からの隔たりを対象とした相対速度テストであれば,LINTREE を使うことも可能です.ただ,私の感触では,LINTREE は逆に精度が高すぎる気がします.
|

|
|
|
@INC (インク) は Perl があらかじめ定義している配列変数です.ここに require などのコマンドがライブラリを探しに行くディレクトリの一覧が保持されています.
Cant'locate micoode.pl in @INC (@INC contains: C:/Perl/lib 〜)
のようなエラーメッセージが出ることがあります.これは micoode.pl が見つからないという意味です.この場合は,
perl -e 'print join ("\n",@INC)'
というコマンドを打つことで,perl が micode.pl を探しているディレクトリのアドレスを確認することができます.
|
|
| マッチ演算子の練習プログラムです. |
use strict;
use warnings;
my $line1 =
"StBack\nMedakaandTetraodon\nTetraodonHuman\nZebra";
my $nameLine = &namePicker($line1);
print "$nameLine\n";
sub namePicker {
my($line1SR) = @_;
my @data = ($line1SR =~ m|(.*?)|gs);
## m||
# /.../ のかわりに m|...| と書くことで,
# スラッシュをエスケープしなくて済む.
## /g
# リストコンテキストで Operator g を使うと,
# マッチを繰り返して ( ) のリストをつくる.
## /s
# メタ文字 . を改行にマッチさせる.
my $line2 = join(',',@data);
$line2 =~ s/\n/,/gs;
return $line2;
}
アウトプットです.
StBack,Medaka,Tetraodon,Tetraodon,Human,Zebra
|
「新版Perl言語プログラミングレッスン入門編」 のサンプルプログラムを参考にしました.こちらです.
|
|
| extractNameLine_perl.tar.gz (2024 年 7 月) |
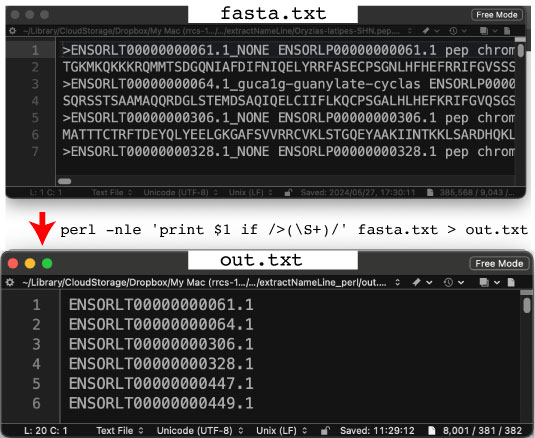 |
-e: 引用符で囲まれた 'perl $1 if />(\S+)/' 部分がスクリプトであることを示す (link)。
\S: 非空白文字 (竹中さんのページ)。
|
|
- .csv で保存すると,自動的に Excel で読めるような設定も可能です.ただし,Mac 側でコマンド+I によって .csv は Excel で読むような設定にする必要があります.
- $text = "gene2234Reanalysis.txt";
$text =~ /gene\d+/;
$group = $&;
によって,$group に gene2234 が入ります.
- $tree = ((MUS15653_mouse,RNOG6076_rat),ENSG157214_human);
@mouseGene = $tree =~ /\w+_mouse/g;
$mouseGene = @mouseGene;
によって,@mouseGene に MUS15653_mouse が入り,$mouseGene に 1 が入ります.
|
|
|
|
|
「駕籠に乗る人担ぐ人そのまた草鞋を作る人」 by A.T.
|
|

























