|
|
|
|
RefSeq は,Reference Sequenceの略で、配列解析に "reference"(リファレンス)となるべき配列データベースのことです.NCBI のスタッフが,最も代表としてふさわしい (参照の基準となる) 遺伝子配列をGenBank などのデータベースから目で見て選んで,RefSeq データベースを作成しています (統合テレビより).
FTP はこちら。
|
|
|
RefSeq と GenBank の違いについては, こちらをご覧ください.重要な部分を抜粋します.
|
|
|
|
GenBank
|
RefSeq
|
|
|
|
研究者自身が投稿
|
NCBI が既存のデータから作成
|
|
同じ遺伝子座から複数のレコードがある
|
主な生物から一つのレコードに限られている
|
|
あらゆる生物 (250,000 種)
|
モデル生物 (4000 種)
|
|
|
|
- In some cases, creation of a RefSeq record involves no more than selecting a single good example from GenBank and making a copy in RefSeq, which credits the GenBank record. In other cases, NCBI in-house staff generates and annotates the records based on the existing primary data, sometimes by combining parts of several GenBank records.
- RefSeq records appear in a similar format as the GenBank records from which they are derived. However, they can be distinguished from GenBank records by their accession prefix, which includes an underscore, and a notation in the “comment” field that indicates the RefSeq status.
|
|
|
ブラウザ:
例1: Branchiostoma belcheri の遺伝子モデル
以下のサイトから、
https://www.ncbi.nlm.nih.gov/datasets/genome/
Branchiostoma belcheli
と入力。 |
 |
その後、
Haploidv18h27
を選択すると、以下が表示されます。
https://www.ncbi.nlm.nih.gov/datasets/genome/GCF_001625305.1/
「Download」を押すとお好みのデータをダウンロードすることができます。
|
例 2: Oikopleura dioica の遺伝子モデルと染色体上の位置
以下のサイトから
https://www.ncbi.nlm.nih.gov/datasets/genome/GCA_907165135.1/ |
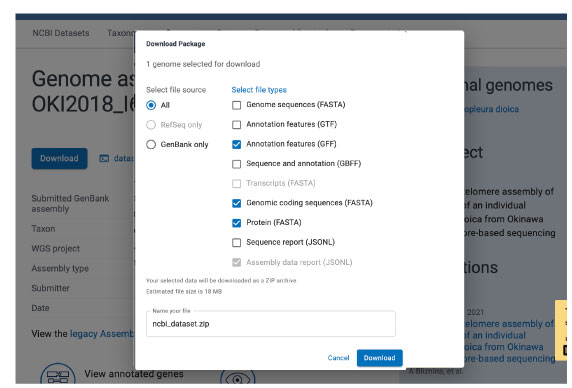 |
| |
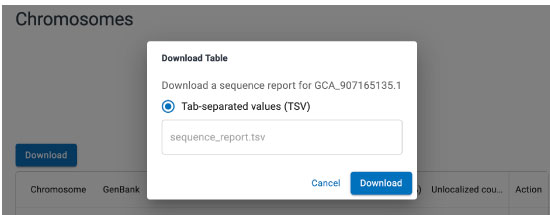
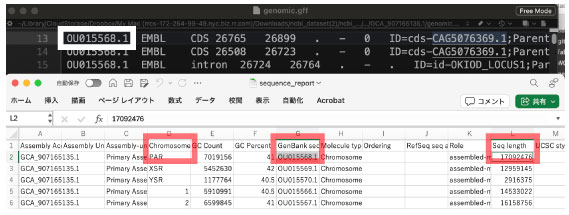
| 染色体 ID に対応する一般的な名称は、sequence_report.tsv (上記のページ下の方にある Downalod からダウンロード)から判定できます。 |
 |
 |
K.T. さんから教えていただきました。ありがとうございます (2024 年 7 月)。
|
ブラウザ ftp:
以上、KH さんから伺いました。ありがとうございます (2023 年 8 月)。
ブラウザ経由の ftp からもデータをダウンロードできます.ブラウザによって動作が異なり、Safari だと「上位のディレクトリへ移動」がありませんが、Firefox にはあります。できないようなら、こちらから直接入ってください。 |
 |
|
ターミナル:
ブラウザを用いてダウンロードする方が速度は早いですが,ターミナルから ftp コマンドを用いてダウンロードもできます.ターミナルから ftp で入ると,60秒の間に何も操作がないと,自動的に接続が切れてしまいます.
junINOUEpro@inouejun-no-MacBook-Pro|~
$ ftp
ftp> open ftp.ncbi.nlm.nih.gov
Connected to ftp.wip.ncbi.nlm.nih.gov.
220-
This warning banner provides privacy and security notices consistent with
....
220 FTP Server ready.
Name (ftp.ncbi.nlm.nih.gov:junINOUEpro): anonymous
331 Anonymous login ok, send your complete email address as your password
Password: [入力せずにリターン]
230 Anonymous access granted, restrictions apply
Remote system type is UNIX.
Using binary mode to transfer files.
ftp> ls
229 Entering Extended Passive Mode (|||50471|)
150 Opening BINARY mode data connection for file list
dr-xr-xr-x 4 ftp anonymous 4096 Oct 28 02:48 1000genomes
-r--r--r-- 1 ftp anonymous 10738466816 Mar 7 2018 10GB
...
dr-xr-xr-x 13 ftp anonymous 253952 Oct 28 02:48 genbank
dr-xr-xr-x 6 ftp anonymous 4096 Feb 28 2018 gene
dr-xr-xr-x 506 ftp anonymous 40960 Oct 28 02:48 genomes
...
dr-xr-xr-x 19 ftp anonymous 4096 Sep 17 19:31 refseq
dr-...
ftp>
....
ftp> pwd
Remote directory: /genomes/all/GCF/000/003/605/GCF_000003605.2_Skow_1.1
ftp> history
0 open ftp.ncbi.nlm.nih.gov
0 cd genomes
0 cd refseq
0 cd invertebrate
0 cd Saccoglossus_kowalevskii
0 cd representative
0 cd GCF_000003605.2_Skow_1.1
0 get GCF_000003605.2_Skow_1.1_rna.gbff.gz
ftp>
こちら「コマンドラインを用いたダウンロード」も参照してください (2018 年 9 月).
|
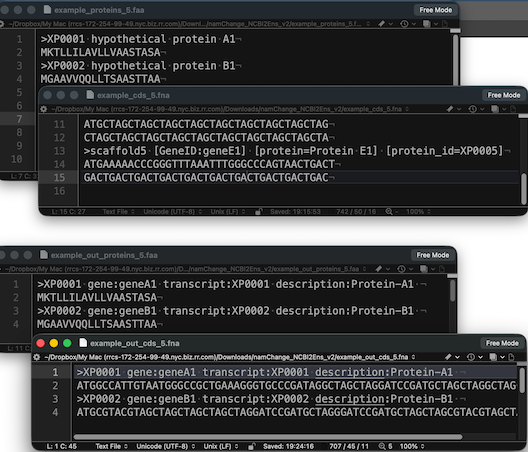
| NCBI 形式の fasta を Ensembl 形式にする |
|
 |
namChange_NCBI2Ens_v2.zip
2025 年 12 月 |
|
 |
namChange_NCBI2Ens.zip
2025 年 11 月
|
ゲノムデータ |
 |
change_NCBI2Ens.tar.gz
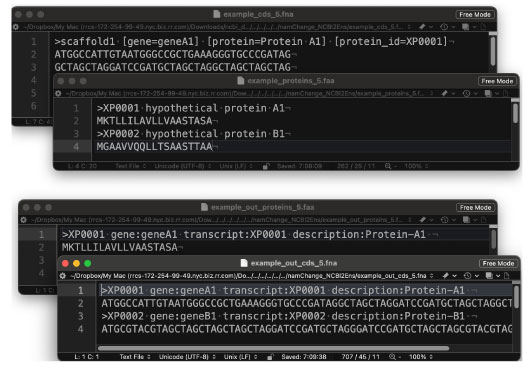
ゲノムデータのファスタファイルの name line を NCBI 形式 (_genomic.fna ファイル) から Ensembl 形式 (.dna.primary_assembly.fa あるいは .dna.toplevel.fa ファイル) に変更します。
繰り返し配列の mask 方は、 以下の記述 (README.txt) を参照。
Example commands to convert lower-case masking to masking with Ns (hard-masked):
perl -pe '/^[^>]/ and $_=~ s/[a-z]/N/g' genomic.fna > genomic.N-masked.fna
-or-
awk '{if(/^[^>]/)gsub(/[a-z]/,"N");print $0}' genomic.fna > genomic.N-masked.fna
(2019 年 10 月)
|
|
 |
retrieve_tss_from_gff.tar.gz
(2020 年 2 月)
|
|
|
|
|
| RefSeq を Local Blast で解析する |
|
They have automated calculation of homologs in HomoloGene:
ftp://ftp.ncbi.nih.gov/pub/HomoloGene
If you want to run local blast, I would suggest the refseq_rna database here:
ftp://ftp.ncbi.nlm.nih.gov/blast/db/
These are pre-formatted for blast. That database contains more than mammals. You can limit your local search of that database to mammals by supplying a text file containing gi list along with the "-l " command line option. Make that gi list in the Entrez Nucleotide database by searching with this query:
mammals[orgn] AND refseq[filter] AND mrna[filter]
Once retrieved on the web page, change Display to "GI List" and send to file.
Get blast here:
ftp://ftp.ncbi.nlm.nih.gov/blast/executables/LATEST
More documentation on setting up and using blast (among other things) is here:
http://www.ncbi.nlm.nih.gov/staff/tao/URLAPI/
|
|








