|
|
|
|
mcmctree はベイズ法に基づいて分岐年代推定を行うソフトウェアです.系統解析ソフトウェアパッケージ PAML に入っています.ここでは Inoue et al. (2010) のデータを例として解析手順を説明します.より簡単な説明はこちらを参照してください.
例題をダウンロードしてください.
mcmctree.example.tar.gz
MCMCTREE で解析を行うには,Windows (XP) であればコマンドプロンプト,Mac (10.6) であればターミナルを用いて,コマンドラインから指令を出す必要があります.このマニュアルでは,ユーザーはコマンドラインの基本的な操作ができるものと仮定して説明を行っています.
現在 MCMCTREE は頻繁にバージョンアップされているので,常にこちらから最新のバージョンをチェックする必要があります.このマニュアルを訳している時点のバージョンは,4.4e です.http://abacus.gene.ucl.ac.uk/software/paml.html.
MCMCTREE では計算スピードを高速化するために,尤度の計算に近似尤度計算 (Approximate Likelihood Calculation) を用いています.近似尤度計算では,尤度関数を近似するために,枝長の最尤推定値 (MLE: maximum likelihood estimate) の多次元正規分布を用いています.実際の解析は,パラメータ (枝長と分散共分散行列) 推定と MCMC アルゴリズムという 2 段階のステップからなります.一方で,近似を行わず MCMC アルゴリズムで同時に配列から尤度を計算する方法も実装されていますが,非常に解析が遅いため非現実的です.近似尤度計算のオプションを選択すると,MCMCTREE は自動的に BASEML や CODEML などパラメータ推定用のプログラムを呼び出します.このため,マニュアルに従ってあらかじめ PATH の設定を行ってください.あるいは,MCMCTREE と同じフォルダに BASEML や CODEML を入れておいても良いです.
専門用語の日本語訳は,できるだけ藤ら (2009) に従いました.このマニュアルは http://abacus.gene.ucl.ac.uk/software/paml.html にある簡易マニュアルに加筆したものです.
|
|
PAML を http://abacus.gene.ucl.ac.uk/software/paml.html からダウンロードします. Windows バージョンはコンパイルせずにそのまま使えます.Windows バージョンは,解凍すると bin というフォルダにアプリケーション (.exe という拡張子がついています) が入っています.
ダウンロードした pamlX.X.tar.gz をダブルクリックし,解凍します (X.X はバージョンの数字です).解凍してできた pamlXX/src に入って,コンパイルを行います.
make -f Makefile
コンパイルを行います.src ディレクトリに baseml, codeml, mcmctree というプログラムが作成されます.mcmctree は baseml や codeml を自動的に呼び出すので,こちらの「Setting up a folder of local programs and change your initialization file for the shell」を参照にパスの設定をしておかないと動かないです.
時間制約付き系藤樹の確認には TreeView を用いると便利です.こちらからダウンロードしてください.
|
|
|
12tr フォルダにある 12tr データセットを用います.12tr_Chi23.phy には,ギンザメ類を中心とした脊椎動物主要系統を代表する 23 種から得られたミトコンドリアゲノム全塩基配列データが保存されています.4 パーティションに区切られており,それぞれ 12 タンパク質遺伝子の第 1, 第 2 座位,および,22 tRNA と 2 rRNA 遺伝子から得られた塩基配列で構成されています.
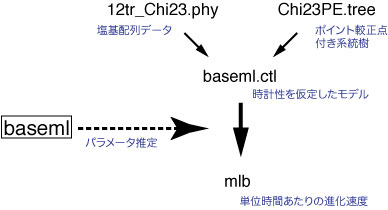
進化速度のラフな推定
まず,データセット全体の進化速度をラフに推定します.この値は MCMC アルゴリズムの事前確立として用います.Mac であればターミナル (Windows であればコマンドプロンプト) から 12tr フォルダに入ってください.mi や BBEdit, TeraPad などのエディターで baseml.ctl ファイルを開けてください.baseml.ctl ファイルは BASEML を動かすためのコントロールファイルです.いろいろなオプションが書いてありますが,詳細は PAML のマニュアルを参照して下さい.ここで BASEML は,時計性を (進化速度が系統間で一定であると) 仮定して,GTR+G モデルを用いた最尤法によって進化速度を推定します.23 種の系統樹は Chi23PE.tree ファイルに保存されています.系統樹の根幹が ‘@4.5’ とラベルされています.ラフな推定なので,ここでは系統樹の根幹が 450 Ma (Million years ago) に分岐したという単純な仮定 (上限や下限制約などを用いない点推定) に基づいて解析を行います.
ターミナルから,
>baseml
と入力して BASEML を走らせてください (‘>’ は入力しないでください).BASEML は自動的に baseml.ctl ファイルを読み込みます.結果は,mlb ファイルに保存されます.mlb ファイルをエディターで開けると,
Substitution rate is per time unit
0.062931 +- 0.002345
という単位時間あたりの進化速度が書かれています.これらの値に基づいて,ベイズ解析で用いる平均進化速度の事前確立を設定します. |
 |
|
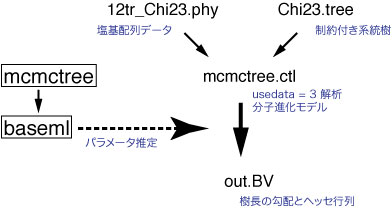
勾配とヘッセ行列の推定
年代推定の最初のステップでは,枝長の勾配 (gradient) とヘッセ (Hessian matrix: 枝長の分散共分散行列) を推定します.勾配 (g) とヘッセ (H) はベクトルと行列として表されます.これらは,枝長の最尤推定値で微分した対数尤度関数 (log likelihood function) の 1 階微分と 2 階微分として得られます.勾配とヘッセは,最尤推定値周辺における対数尤度表面の形を表しており,テイラー展開を用いた対数尤度曲線の近似にも用いることが可能です (dos Reis and Yang 2011).g と H は時計性を仮定しないで得られた無根樹から推定されます.
エディターで mcmctree.ctl ファイルを開いてください.これは MCMCTREE を動かすコントロールファイルです.解析に用いる配列は 12tr_Chi23.phy に,系統樹は Chi23.tree にそれぞれ保存されています.系統樹は rooting されている必要があります.枝長は書かれていません.そして,化石記録に基づく年代制約が系統樹に書かれています (例えば ‘B(4.22,4.63)’ です).制約の設定方法はマニュアルを参照してください.なお,この段階で化石制約は解析に用いられません.
mcmctree.ctl ファイルに,
usedata = 3
と書かれています.これは,MCMCTREE に枝長の最尤推定値と g, H, を推定させるオプションです.まだ年代推定は行われません.ターミナル上で 12tr フォルダにいることを確認し,
> mcmctree
と入力すれば,MCMCTREE による解析が始まります.MCMCTREE は,系統樹を無根にして,一時的なファイルに保存します.そして BASEML が呼び出されて,時計性を仮定しないで枝長が最尤推定され,g と H が得られます.
データは 4 パーティションに区切られているため,BASEML は枝長,g ベクトル,H 行列を 4 セット推定します.結果は out.BV ファイルに保存されます.out.BV ファイルを開くと,最初の行には 1 番目のパーティションで解析に用いられた種数が書かれています.続く行には,それぞれ,枝長付きの系統樹,枝長 (系統樹から抜き出した数字),勾配 (枝長 0 の枝が無ければ,すべての勾配は 0 か 0 に近い値になって,正常な解析結果が得られます),そして最後にヘッセ行列が書かれています.以上が 4 パーティション分書かれます.out.BV ファイルの名前を in.BV に変更してください.
例えば長大な配列データを解析するときなど,クラスター等を活用して上記の推定値をパーティションごとに計算させることも可能です.この場合は 4 パーティションを 4 つの配列ファイルに分けて 4 つのフォルダー別々に保存するなどの処置が必要です.baseml.ctl ファイルもパーティションごとに分けて作成して,直接 BASEML を別々に走らせます.この場合,解析に用いる系統樹が無根である必要があります.有根樹を無根にするにはMCMCTREE を usedata=3 オプションで一時的に走らせて,tmp1.trees 等に保存される無根樹を解析に用いると良いでしょう.勾配とヘッセは rst2 ファイルに保存されます.パーティションごとに得られた rst2 ファイルを一つのファイルにまとめ,in.BV ファイルとして保存します. |
 |
|
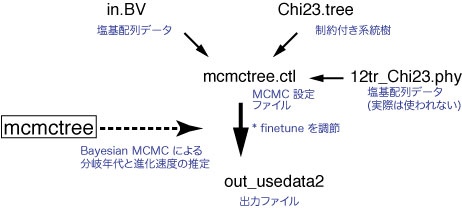
近似尤度計算を用いた分岐年代の推定
mcmctree.ctl 開いて usedata のラインを
usedata = 2
と変更してください.このオプションによって近似尤度計算を用いた MCMC アルゴリズムを行います.
outfile = out_usedata2
とすれば,usedata=3 の解析結果とは別のファイルに結果を保存することができます.
rgene_gamma は,系統樹全体の進化速度を設定するための事前ガンマ分布のパラメータです.事前ガンマ分布の形は,形状パラメータ (a) と 尺度パラメータ (b) によって決まります.平均を m,標準偏差を s とすると,a,b,m,s の関係は以下の式で表すことができます:
a = (m/s) 2
b = m/s2.
BASEML を用いたラフな推定で得られた進化速度の値から m = 0.06 として,同様に s = 0.06 とします.すると形状パラメータ a は,
a = (0.06/0.06) 2
= 1,
となり,尺度パラメータ b は,
b = 0.06/0.062
=16.6666
となります.このためコントロールファイルでは,
rgene_gamma = 1 16.7
と設定します.形状パラメータが 1 のとき,ガンマ分布は指数分布になります.上記のパラメータでは,ガンマ分布は拡散した分布となります.統計パッケージ R を用いて事前分布を確認することができます.R のコンソールに
> curve(dgamma(x, 1, 16.7), from=0, to=0.2)
と入力すると,事前分布の形を見ることができます.
次に,進化速度の分散を設定する事前確立を指定します.このパラメータは枝間で進化速度にどれほどバラツキがあるのか設定します.ここでは,進化速度は対数正規分布に従うと仮定しています.mcmctree.ctl ファイルで,
sigma2_gamma = 1 4.5
と設定してください.これらの値は,データによって変更してください.
最後に MCMC サンプリングに使われるバーンインとサンプル数を設定します.
burnin = 50000
nsample = 10000
mcmctree を走らせます.
> mcmctree
MCMCTREE は近似尤度計算を行うにあたり,アライメントファイル,化石制約が施された tree ファイル,in.BV ファイルを読み込みます.MCMC アルゴリズムを走らせたらスクリーンアウトに出てくる採択率 (acceptance proportion) を見てください.これらは 30% 前後の値 (20〜40%が良いですが,15〜70%でも解析可能) が理想的です.もし採択率がこの範囲内でない場合は,Ctl+C によって解析を中断し,finetune parameters を調節して再度解析をスタートさせてください.調節方法はマニュアルをご覧下さい.なおバージョン 44e から,finetune の設定を自動的に行うオプションがつきました (0: があると,数値がすべて 1 になってしまいました.0: をとったら正常な値が出てきました [2011 年 9 月]).以下の行にあるコロンの前の数字を 1 にすると自動的に適切な finetune を探すようですが,まだ充分にテストされていないようです.詳しくはマニュアルをご覧下さい.
finetune = 0: 0.04 0.2 0.3 0.1 0.3
結果は out_usedata2 ファイルに保存されます.ここには,それぞれの分岐の推定年代と 95% 信頼区間 (CI) も書かれています.生データは mcmc.out ファイルに保存されており, Tracer (分岐年代推定プログラム BEAST に含まれています) かあるいは R の CODA パッケージで解析することができます.
|
|
|
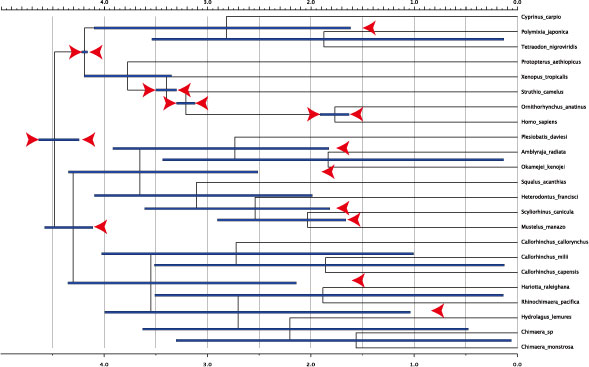
usedata = 0
として解析すると,配列データを用いない解析を行うことができます.この場合,sampfreq を 10 倍多くして,
sampfreq = 500
として解析を行ってください.得られた結果は下図のようになりました.矢印は制約です.
この解析によって,
1. tree file に施した化石制約に矛盾はないか.
2. Posterior (配列を用いた解析の結果) と比較することで,配列データがどれぐらい結果に影響を及ぼしているか.
がわかります. |
|
|
|
アミノ酸配列アライメントである AA_Chi23.phy を解析します.AA フォルダに入ってください.ここではパーティションを切らない解析を行っています.ここで用いるアミノ酸配列データは,塩基配列データの解析で用いた第 1,第 2 座位に対応するものです.
手順は塩基配列データの解析と同じです.アミノ酸配列を解析するので,進化速度をラフに推定するために,BASEML の代わりに CODEML が必要です.その後,MCMCTREE は CODEML を呼び出し,枝長の最尤推定値,g,および H,を推定します.mtREV24.dat ファイルを PAML パッケージにある dat フォルダから AA フォルダにコピーしてください.
> codeml
と入力して,CODEML を走らせます.CODEML は時計性を仮定して,アミノ酸配列データセットを最尤法によって解析します.アウトファイルにある,
Substitution rate is per time unit
0.047131
という推定進化速度を見つけてください.
mcmctree.ctl ファイルの内容を適宜変更し,時計性を仮定しないで枝長および g,H を推定します.mcmctree.ctl ファイルで
seqtype = 2
が選択されているか確認して下さい.このオプションによって MCMCTREE はアミノ酸配列データを解析します.
> mcmctree
と入力し,MCMCTREE を走らせます.
塩基配列データの解析と同様に,MCMCTREE は近似尤度計算に必要な各種のファイルを自動的に作成し,CODEML を呼び出して解析を始めます.out.BV ファイルが作成され,枝長 , g, H, が保存されます.out.BV ファイルを in.BV という名前に変更してください.mcmctree.ctl ファイルの内容も変更してください.例えば,時計性を仮定して推定した進化速度を参考に,進化速度の事前確立を設定してください.その後,MCMC アルゴリズムを実行します.
|
|
近似尤度計算で解析を行う利点として,混合したデータセットを解析できる点があげられます.例えば,塩基配列とアミノ酸配列をパーティションに分けた解析が可能です.枝長,g,H が in.BV ファイルに保存されていれば,MCMC アルゴリズム (usedata=2) ではアライメントデータを実際の解析に用いることなしに年代が推定されます.
ここでは例として,アミノ酸配列データと tRNA,rRNA 遺伝子の塩基配列データをつなげた解析を行います (最初がアミノ酸配列のパーティションです).in.BV ファイルを 12tr フォルダから AAtr フォルダにコピーしてください.in.BV ファイルにある最初の 2 パーティションはコドンの第 1,第 2 座位から得られたパラメータであるため削除します (tree,枝長ベクトル,勾配ベクトル,ヘッセ行列 2 セット分).AA フォルダに入っている in.BV ファイルの内容をすべてコピーして,AAtr フォルダに新たに作成した in.BV ファイルの最初の部分に貼付けます.これで in.BV ファイルには,アミノ酸配列データ,tRNA および rRNA 遺伝子の塩基配列データから得られた 3 パーティション分のパラメータセットが保存されることになります.
配列ファイルも 3 パーティション用に設定する必要があります (12tr_Chi23_P3.phy).MCMC アルゴリズムの際に配列データはチェックされますが,使われることはありません.配列ファイルにあるそれぞれのパーティションは,in.BV の対応するパラメータセットと同じ種数である必要があります.in.BV と配列データーの種数が対応しているならば,例えば,20 種から得られた tRNA 遺伝子の塩基配列データと 30 種から得られたアミノ酸配列データを解析することも可能です (訳者はやったことがありません).この場合,化石制約の施されたマスター tree は全部の種を含めた 30 種からなる必要があります.MCMCTREE は配列アライメントファイルの種数をチェックします.ここで注意したいのが,配列データファイルは,アミノ酸配列かあるいは塩基配列どちらか一種類である点です.両者をまぜた配列データを作成してはいけません.その代わりに,それぞれのデータタイプにあわせて BASEML か CODEML の解析を別々に行った後で,異なるパーティションの枝長,勾配,ヘッセ行列をつなぎ合わせます.このため,例えば,ある枝長とヘッセ行列は塩基配列から得られ,またあるものはアミノ酸配列,あるものはコドン配列 (訳者はコドン解析をやったことがありません) から得られることになります.言わば,実際には異なる種類のデータから別々に in.BV ファイルのパラメータを得ているにも関わらず,MCMCTREE をだまして同じ種類のデータセットを解析しているように見せかけていることになります.
AAtr フォルダにある mcmctree.ctl ファイルを編集します.速度事前確立を適宜変更してください.sigma2 の事前分布も調節してください.
事後分布が収束するかどうか確認するために,それぞれの MCMC アルゴリズム (usedata=2 の解析だけ) は最低でも二回行ってください.この簡易マニュアルでは,MCMC アルゴリズムは少なくとも 6 回行ったことになります.同じコントロールファイルを使った 2 回の MCMC アルゴリズムで大きく違った値が得られた場合は,収束に至っていないと考えられます.この場合は,より多い回数で MCMC アルゴリズムを行う必要があります (コントロールファイルにある sampfreq オプションの値を増やします).これにあわせてバーンインを増やす必要もあります.finetune も変化させる必要が出てくるでしょう.
|
|
|
|
Inoue, J.G., Miya, M., Lam, K., Tay B.H., Danks, J.A., Bell, J., Walker, T.I., Venkatesh, B. 2010. Evolutionary origin and phylogeny of the modern holocephalans (Chondrichthyes: Chimaeriformes): a mitogenomic perspective. Molecular Biology and Evolution, 27: 2576- 2586.
dos Reis, M. and Yang Z. 2011. Approximate likelihood calculation on a phylogeny for Bayesian estimation of divergence times. Molecular Biology and Evolution. 28:2161–2172..
Yang, Z. 2009. 分子系統学への統計的アプローチ・計算分子進化学.藤博幸, 加藤和貴, 大安裕美,訳.東京,共立出版.
Yang, Z. (2007). PAML 4: Phylogenetic Analysis by Maximum Likelihood. Mol. Biol. Evol. 24: 1586–1591.
|
|
|
|