|
|||
|
|||
| MEGA は,配列のエディット,アライメント,系統関係や時間軸付き系統樹の推定,などを行うソフトウェアです.GUI バージョンと CUI バージョン (megacc) があります。 |
|||
|
|||
|
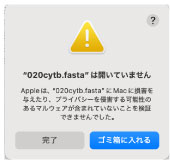
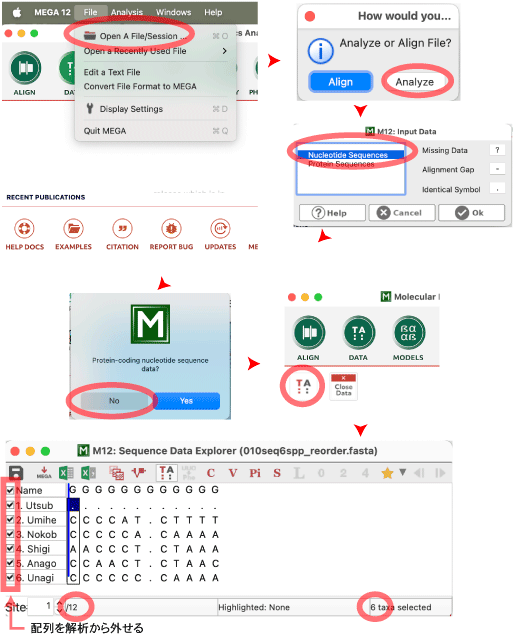
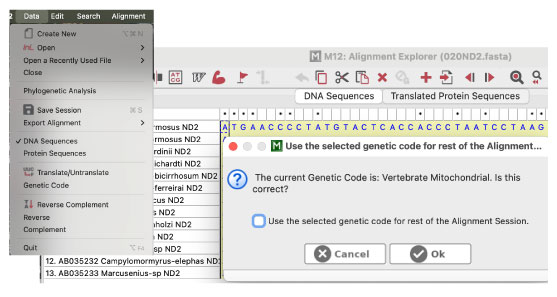
DNA ファスタファイルを読んで、翻訳、アライメント、異なるアミノ酸サイトのカウントを行います。ここでは、Mac, MEGA 12 (64-bit) ALPHA を使いました。mac でインフォマティクスを参照しました。 fasta ファイルの読み込み なお Mac では、拡張子が .fasta のファイル (上記 test.fasta など) をエディタで開こうとすると、下右のようなメッセージが出てしまいます。vim では問題なく開けました。 |
|||
  |
|||
|
|||
 |
|||
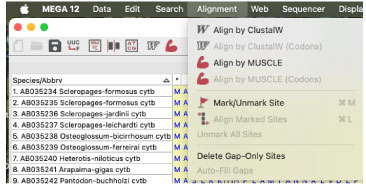
アライメント Alignment > Align by ClustalW などを選びます。 |
|||
 |
|||
| 翻訳 Translated Protein Sequences を選びます。遺伝暗号は、Data メニューから Genetic Code を選択し、「Vertebrate mitochondrial」などを選択します。このとき、コマンド + A で配列を全て選択しておく必要があります。 |
|||
 |
|||
アライメントの編集 |
|||
 |
|||
| 私の理解では、DNA やアミノ酸を異なるものに変えたり、種名を変更する場合は、一度、fasta 形式に変換して (test_AA.fas) 行う必要があるようです。 | |||
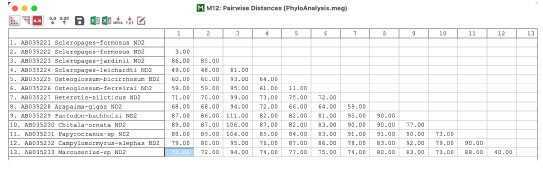
| 異なるサイトのカウント "Molecular Evolutionary Genetics Analysis" パネル > DISTANCE > Compute PairwiseDIstances... を選びます。 |
|||
 |
|||
異なるサイトを数えるだけのオプション, No. of differences を選びます。 |
|||
 |
|||
 |
|||
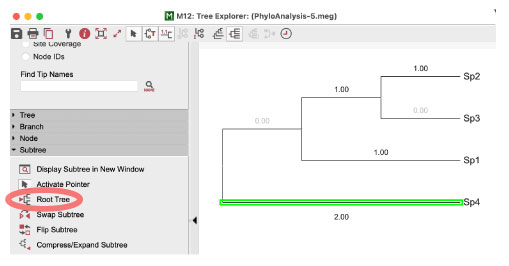
| ルートを変更する | |||
 |
|||
2025 年 5 月
|
|||
|
|||
ここでは,Mac のコマンドライン (CC) バージョンを説明します.スライドによる説明も参考にしてください.Documentation はこちらです. Mac の mega-cc v11 では、cluster アライメントは動きましたが、muscle アライメントは動きませんでした。 megacc (CUI) バージョンは、.mao ファイルをあらかじめ GUI などで作成して解析に用います。 |
|||
 |
|||
次に、作成した .mao ファイルを使って、megacc を動かします。
muscle アライメントはエラーが出ました (2023 年 8 月)。
|
|||
|
|||
010infer_NJ_protein.mao
010aaseq.fas
020_out
020_out.nwk
まだ充分に確かめたわけではありませんが,NJ 解析ではアウトグループを設定できないようです.おそらく,解析を行った後で,rooting を改めてやることになると思います.また,アミノ酸解析の経験的モデルは, JTT しか対応していません. |
|||
|
|||
| Mega 7 を使って説明します.mega_reltime_example.tar.gz をダウンロードしてください.Mega 11 でもこの例題を解くことができました。MEGA7 より Mac で操作しやすくなっていました (2021 年 10 月)。
|
|||
 |
|||
=> Data > Open A File/Session から,Vertebrate6.fas を選んでください.fasta 形式だと,アライメントファイルの読み込みがスムーズです.Phylip 形式だと,面倒です.その後,以下のように選んでください. "How would you like to open this fasta file" |
|||
 |
|||
=> Clocks > Compute Timetree (ML) |
|||
 |
|||
| Step 1: Load Seqence Data すでに選んだので,何もやらなくて良いです. |
|||
Step 2: Load Tree File |
|||
| Step 3: Specify Outgroup Select Branch... => |
|||
 |
|||
| Please confirm: .... => Yes |
|||
| Step 4: Specify Calibrations Add Constraints... |
|||
 |
|||
| 上記の制約を,以下の画面を参考に入力してください. | |||
 |
|||
| 上記の制約は Calibrations Export ファイルに入っています. File > Import Calibration Data からこのファイルを選ぶことができます. |
|||
| Setp5: Choose Analysis Settings Set Options... |
|||
 |
|||
| 最後に,Execute を押して,解析を走らせます. | |||
 |
|||
| 以下のような結果が得られるはずです. | |||
 |
|||
こちら「RelTime による分岐時間推定」を参照しました. |
|||
| 推定された時間軸付き系統樹や時間制約、解析設定の要約などを保存できます。以下の画面は MEGA11 です。 | |||
|
|||
| なお、各分岐に推定された年代は、私の場合、Newick format を FigTree で開き、Node Labels > Display: Node ages で得ています (2021 年 10 月)。 | |||
|
|
|||
Confidence Interval MEGA では、estimated error bar が confidence interval に相当するようです (Mello et all 2018, P2339右下)。 RelTime-ML 解析を行った後で、Divergece Times のチェックボックスを入れて、Show Node Height error Bars (2SE) をチェックすると、画面にエラーバーが保存されます (下図)。 CI の推定値は、File > Export Timetree (Tabular) を選ぶと、.xlsx ファイルとして保存されます。このファイルがどこに出力されるのかよくわからないですが、Excel で自動的に開かれるので、任意の場所に保存できます (2022 年 1 月)。 |
|||

|
|||
|
|||
こちらのビデオが参考になります.
|
|||
|
|||
Mello B, Tao Q, Tamura K, & Kumar S (2017)
|
|||
|
|

