概説:
-
phyloBARCODER は、系統樹を推定してメタバーコーディング DNA 配列の種判定を行うウェブツールです。種名を判定し、リード配列から同じ種の配列を選定します。 データベースは真核生物に特化しており、ミトコンドリアゲノム全遺伝子配列からなります。
インプットファイルとして、環境 DNA やメタバーコーディングサンプルから得られた配列一群 (ここでは anonymous 配列と呼びます) をアップロードして用います。 データベースとして、ユーザー独自が作成した参照配列も利用できます。 期待される結果として、BLAST identity が同じ場合でも、より近い配列を選定することがあります(result674_Tree_vs_similarity.zip)。

(A) Species Identification
-
ここでは例として、12S rRNA 環境 DNA 配列 (MiFish) をアップロードして用います (Yu et
al. 2022)。
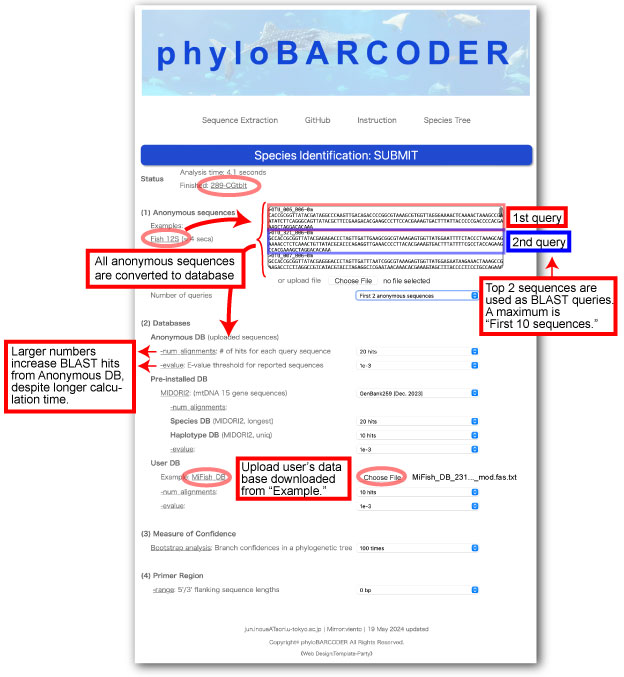
ユーザーは、Fish 12S と書かれたリンクから、これら anonymous (種名のわからない) 環境 DNA 配列を、テキストボックスにコピー&ペースします。ここでは、2 つの OTU を同時に種判定するために、Number of queries を Fist 2 sequences にセットします。
- Anonymous sequences
- KS1815-B06-0m_ASV.fasta.txt
- Raw data
- KS1815-B06-0m_S50_R1_001.fastq.gz
- KS1815-B06-0m_S50_R2_001.fastq.gz
- (Yu et al. 2021) Example: Copepod analysis
- Reference sequence
- Metridia_pacifica_lucens_MZGdb_Selected.txt
- (MetaZooGene Atlas & Database, 11 Oct 2023)
Number of queries: このボックスは、「First 10 sequences」 (fasta 配列トップに記載した10 配列) を最大としています。「First 2 sequences」を選んだ場合、BLAST search ではこれら 2 つの配列だけが用いられます。しかし、2 クエリ配列を含んだ全ての anonymous 配列が、「Anonymous DB」に変換されます。このため、fist 2 sequences と類似した、アップロードした anonymous 配列は、その後作成される多重配列に含まれます。残りの、anonymous 配列 (BLAST で選ばれなかった) は、アライメントに含まれません。
-num_alignments と -evalue オプション:アライメントに含まれる配列数を調節することで、より正確な種判定を可能にします。
Anonymous DB では、これらの数値は、BLAST hits の数を増加または減少させることで、クエリ配列と同じグループに属する anonymous 配列を明らかにします。
Species DB (Pre-installe DB) では、系統樹内部で、注目する種あるいはグループ判定のために用いる適切なルート配列を選定します。Haplotype DB では、同じ種から得られた異なる参照配列 (haplotypes) を加えることで、種、グループあるいは集団の境界を鮮明にします。
User DB では、このデータベースからアライメントに加える配列数を増減します。もし -num_alignments を 0 にすれば、そのデータベースは利用されません。
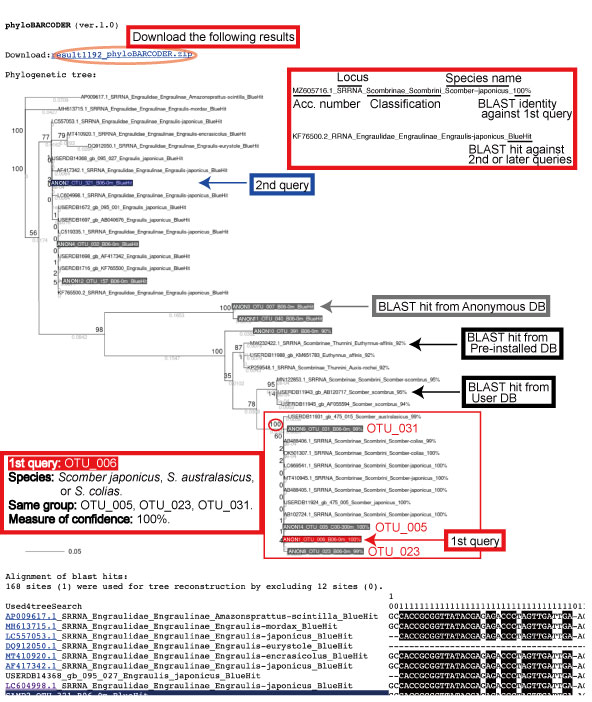
結果は、Status > Finished の隣にあるリンクを押すことで得られます
ユーザーは、得られた系統樹を直接目でみて、種判定を確認する必要があります。
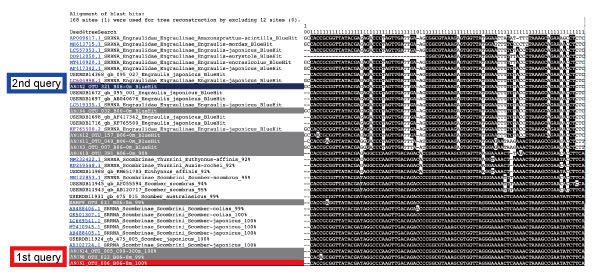
種判別の確認に、アライメントも利用できます。
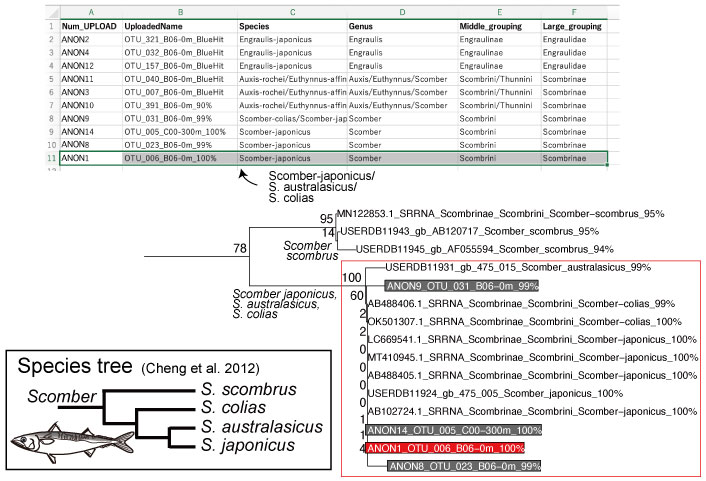
phyloBARCODER は、ユーザーが設定したクエリ配列の種判定結果を種リストとして産出します。種と分類は、Pre-installed DB から得られたBLAST hits* から作成され、taxon_assignment_tree.csv ファイルに保存されます。上の例では、OTU_6 の種名は、Scomber japonicus だけでなく S. australasicus と S. colias も候補として挙げられています。より種名を絞るには、それぞれの種の分布を考える必要があります。
*User DB (ユーザーが独自に作成した参照配列) から得られた BLAST hits からは、種名は作成されません。
Example: Fish ASV analysis
テキストボックスの配列だけで系統樹を作成できます。
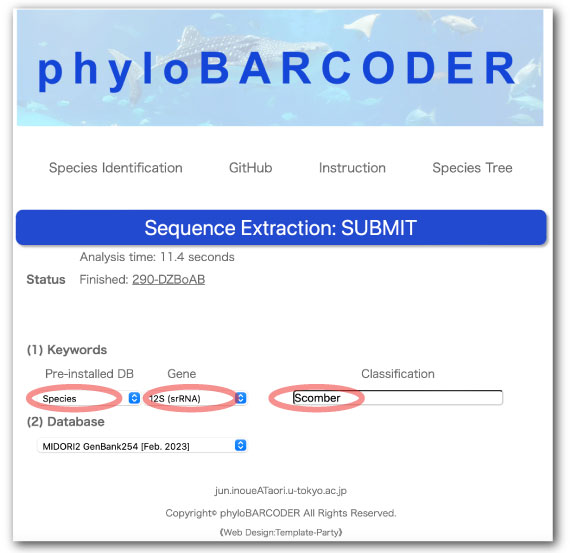
(B) Sequence Extraction
-
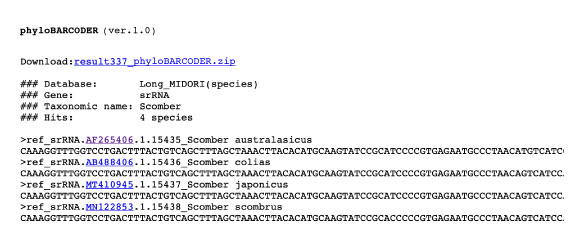
Scomber 属で、12S rRNA 遺伝子の参照配列の登録がある種を数えます。
以下のパラメータを入力あるいは選んでください:
- Pre-installed DB
- Species
- Gene
- 12S (srRNA)
- Classification
- Scomber
Citation
-
Inoue J. et al.
phyloBARCODER: A web tool for phylogenetic classification of eukaryote metabarcodes using custom reference databases. Molecular Biology and Evolution 2024, 42: msae111. doi: 10.1093/molbev/msae111